Nemegyszer előfordult, hogy biológusok, orvosok tanácsot kértek tőle, hogyan végezzenek el egy bizonyos elemzést. Régebben elmondtam a program nevét, felsoroltam a paramétereket, amikor váratlanul megkérdezték
- Tudni fogom, hova kell klikkelni?
- Persze. Sehova. Ez egy parancssoros program.
Ekkor derül ki, hogy a kérdező valami Windowsos programot szeretne, ahol csak klikkel egyet és megkapja az eredményt. Mindezt lehetőleg egy átlagon aluli teljesítményt nyújtó számítógépen. Ilyenkor természetesen nem tudok érdemben segíteni, legfeljebb felajánlom, hogy majd lefuttatom én a programokat egy GNU/Linuxos gépen.
Később már visszakérdeztem, milyen számítógépeken akarnak bioinformatikát művelni, amivel a félreértéseket elkerülhettem. Azért felmerült bennem, mégis milyen lehetőségek vannak Windowson? Ebben a posztban kipróbálok néhányat ellenséges operációs rendszeren.
Tesztkörnyezet
A tesztgép egy AMD 4400-as 2,2GHz-en, 4GB memóriával. Az operációs rendszer 64 bites Windows 7, amire a cikk idején az összes automatikus frissítés felment. A webes megoldásokkal nem foglalkoztam, csak olyan programok próbaverzióit használtam, amit bárki le tud tölteni. (Mint az látszani fog a posztból, ez nem is mindig olyan egyszerű)
A tesztfeladat elég egyszerű: Egy Ion Torrent szekvenciát kell a humán referenciához illeszteni, megkeresni a variációkat, és összevetni egy már ismert eredménnyel. A szekvencia nyilvánosan elérhető, de sajnos regisztrálni kell az oldalra. A szekvenáláshoz egy AmpliSeq kitet használtak, ami több, daganatos megbetegedésekben szerepet játszó genetikai polimorfizmusok környékére szűkíti a szekvenálást. Az oldalon a szekvencia mellett megtaláljuk a szekvencia célpontokat (BED állomány), valamint egy másik analízis eredményét (VCF).
Geneious 5.6.4
A Geneious 14 napos próbaverziója könnyen letölthető. Még valódi e-mailt sem kell megadni. A .gz kiterjesztésű fájlokat elkezdte betölteni, de egy idő után rájött, hogy nem tudja, mit is tartalmaz, ezért nem ment tovább. A kibontás után, importálás közben automatikusan meghatározza a fájl típusát. A referencia fájlokkal könnyedén boldogult, még arra is rákérdezett, hogy a sok kijelölt FASTA állományt egybe kezelje-e.
Az illesztésnél már adódtak problémák. A teljes humán referencia futtatásához 8Gb-ra van szükség, holott minden egyes kromoszóma külön-külön beleférne 4Gb-ba. Az alapértelmezett memória, amit a program fel tud használni 1Gb, amit nekünk kell manuálisan megnövelni. (De ehhez adminisztrátori módban kell futtatni a programot.)
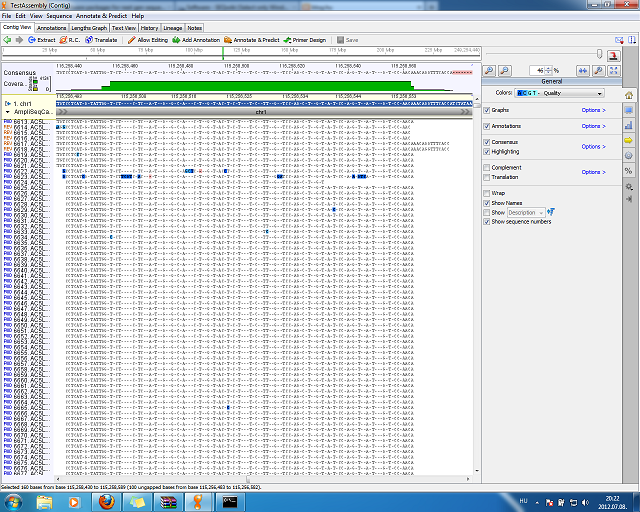
A BED állományt is szépen beolvasta, de előtte ki kellett jelölni, hogy melyik referenciát kívánjuk annotálni. Ez általánosságban jellemző a programra, hogy először mindig az adatokat kell kijelölni, és csak utána kell kiválasztani a funkciót.
Az illesztés megtekintésekor ellenben speciális menüket kapunk, hogy a további vizsgálatokat elvégezhessük itt. A nukleotid polimorfizmusok keresését a kissé megtévesztő Annotate & Predict menüpont alatt találjuk. Az eredményeket pedig CSV állományba menthetjük. Nem VCF, de azért megteszi. Az viszont már gond, hogy exportáláskor az összes annotáció egyszerre kerül mentésre, tehát a variációk és az amplifikált pozíciók ugyan abban a fájlban lesznek. Az illesztés is exportálható SAM formátumban.
Mivel a VCF-t nem tudtam importálni, ezért nem tudtam összevetni sem, amit kaptam a teszt adatokkal. Az amplifikált régióban található variációkat is csak szemmel tudtam azonosítani, ráadásul az exportálás során összemossa az annotációkat. Ugyanakkor pozitívum, hogy a táblázat elemei között klikkelve az illesztés megfelelő pozíciójára ugrik, ami segít.
Összességében ez a program elég sok manuális munkát ad. Nehézkes, az egyes funkciók eléréséhez felugró ablakok környezetfüggő menüiben kell matatni. Továbbra is az a véleményem, hogy klasszikus feladatokhoz remekül használható, de újgenerációs szekvenálások esetén inkább kerüljük el.
CLC Genomics Workbench 5.5 beta 2
Úgy döntöttem, mivel nemsokára kijön az új verzió, ezért a bétával fogom a teszteket lefuttatni. Kérnek regisztrációt, de semmi valódi adatot nem kell megadni.
A referencia importálása kromoszómánként történt, és a program kromoszómánként is kezeli őket. Nincs olyan lehetőségünk, egy egybe fogjuk őket, mint a Geneious esetén. Természetesen egy könyvtárba szervezhetjük őket, de az mégsem ugyan az. A FASTA állományok importálásával nem volt semmi gond, leszámítva, hogy sokáig tartott.
A readeket szekvenáló gép alapján lehet importálni, az Ion Torrent támogatott, nem volt vele probléma. Az annotációval annál inkább. Sem BED, sem VCF állományokat nem tudtam betölteni, de a GFF támogatott.
Illesztésnél megadhatunk több referencia fájlt, és nem akarja egyben betölteni az összeset a memóriába. A beállítások egyszerűek egy parancssoros programhoz képest. Bár most nem használtam, lehetőség van egyszerűbb kötegelésre is, ha több szekvenciát is szeretnénk hasonló beállításokkal futtatni. Nem túl bonyolult, de adott esetben hasznos is lehet. A kész illesztést elmenthetjük SAM/BAM formátumban.
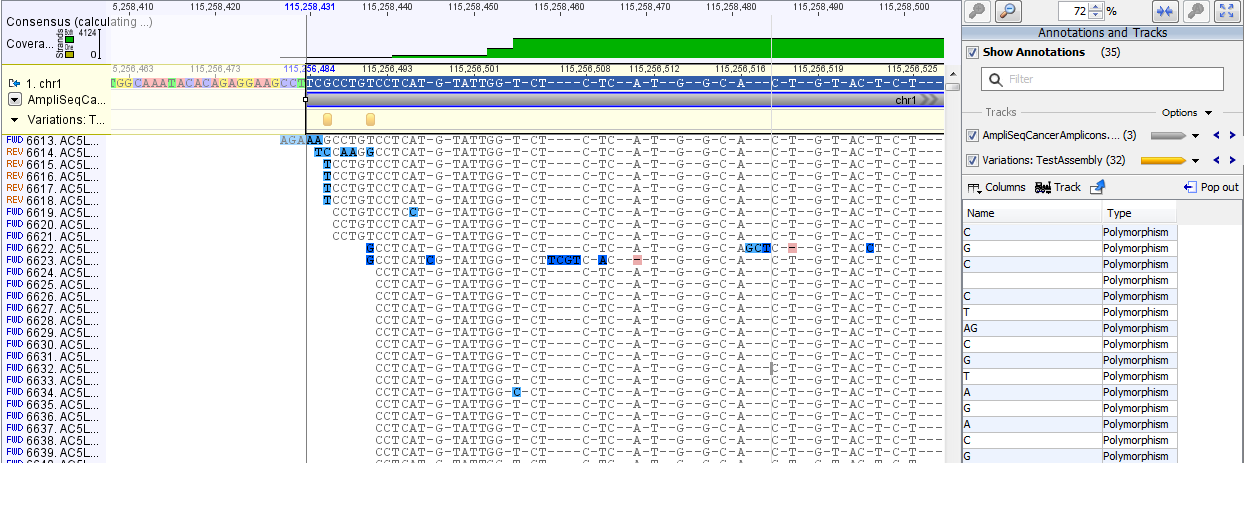
A variációk felderítésére két módszer is van. Az egyik a nemrég debütált Probabilistic Variant Detection, míg a másik, a hagyományos alapokra (a referenciától eltérő readek arányán alapul) épülő Quality-based Variant Detection. Az Ion Torrentre jellemző homopolimer hibát képesek az algoritmusok figyelembe venni. Az eredmény mindkét esetben lehet annotációként a referenciára rakni, vagy külön táblázatban nézegetni. A Geneiousnál megszokott klikkeléssel nem lehet a referencia megfelelő pontjára ugrani, azt kézzel kell kimásolni (a vágólapot sem használhatjuk a koordináták másolására). A táblázatot CSV formátumban is elmenthetjük.
Az eredményt annotációs trackként is megjeleníthetjük. Ez, bár nem egy szofisztikált eszköz, de használhatjuk a variációk összehasonlítására. Mivel VCF-t nem tudtam betölteni, ezért a kétféle variáció detektáló algoritmus eredményét hasonlítottam össze a képen. Az elemzésben több segítséget nem kapunk.
A CLC népszerű újgenerációs szekvenálások feldolgozásában. Rengeteg eszközt ad a kutatók kezébe, ha kis számú adattal kell szöszmötölni. A számítógép mellé persze nem árt egy hagyományos jegyzetfüzet, mert a vágólap használata nélkül kénytelenek vagyunk kézzel lejegyezni apró információkat.
GeneSpring NGS 12.1
A GeneSpring, mikor még aktívan használtam, egy chip elemző program volt csupán. Azóta ők is nyitottak az NGS felé. A próbaverziót regisztráció után lehet letölteni, ahol bármilyen e-mail címet megadhatunk. Mivel már korábbról volt hozzáférésem, ezért könnyedén le is tudtam tölteni a próbaverziót, amit 20 napig használhatunk.
Sajnos a licencet már nem sikerült szereznem hozzá, mert 6 évvel ezelőtt letöltöttem egy másik próbaverziót, ezzel eljátszottam minden életemet. Később sikerült aktualizálni a licencet, így már nyugodtan kipróbálhattam a programot.
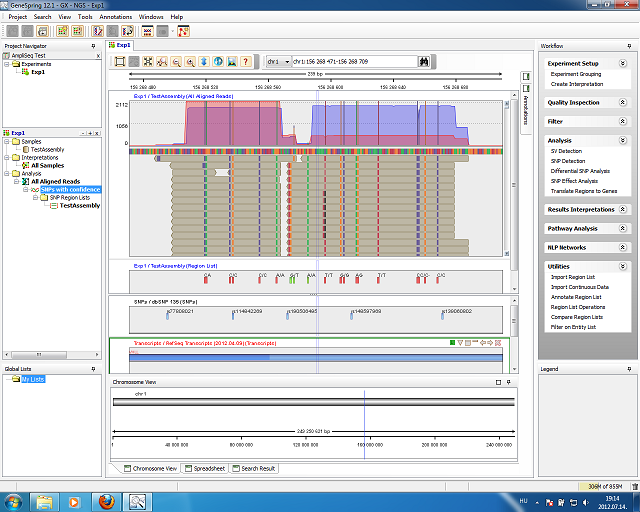
Az első kellemes meglepetés az annotációk automatikus frissítése volt. Az első indítás után kiválaszthattuk az általunk vizsgált lény referenciáit és beállíthatjuk, hogy mit töltsön le. A szokásos referencia importálást ezért kihagytam és a frissítést választottam. Kiválasztottam a dbSNP-t is, ami jelentősen megnövelte a letöltési időt. Nagyjából 4 órán át tartott a RefSeq, a genom és a 135-ös dbSNP importálása, amit én soknak tartok.
Fontos kihangsúlyozni, hogy a Genesprint az Agilent terméke, ami azért fontos, mert a szokásos DNS chipek mellett más laboratóriumi kiteket is forgalmaznak. Ezen kitek kiértékeléséhez szükséges egyéb adatok a programba be vannak építve. Például a SureSelect targetáló termékcsalád genomi célpontjai.
A program nem tartalmaz short read import funkciót. Egyáltalán semmit nem tudunk kezdeni short readjeinkkel ezzel a programmal. Az illesztés ki-ki oldja meg maga saját eszközével. A BAM fájlt már be lehet tölteni és lehet keresni nukleotid polimorfizmusokat.
Sajnos nem találtam meg a módját, hogyan lehetne VCF fájlokat beimportálni, pedig ha sikerült volna, akkor a Differential SNP Analysis segítségével izgalmas összehasonlításokat lehetett volna csinálni. De ha BED vagy CSV formátumban vannak adataink, akkor mindez nem jelent problémát.
Egyetlen komoly problémám volt csak a programmal, hogy rettenetesen lassú volt. Minden egyes művelet esetén valamit nagyon töltött. Még ha csak a látható ablakokat akartam kimenteni a program beépített képlopó módszerével, akkor is elkezdett tölteni, frissíteni. Ha kicsit átméreteztem az ablakokat, akkor is elkezdett újrarajzolni mindent, amihez töltött valamit a merevlemezről. Az a gyanúm, hogy semmilyen indexelést nem végez a program, ezért minden újrarajzolás idején elkezdi betölteni a referenciát és az annotációt, hogy megkeresse a kirajzolandó adatokat.
A program sokat tud, de nehéz eligazodni rajta. A súgó pedig leírja, hogy miként kell egy analízist elvégezni, de a hibakezelés teljes egészében hiányzik. Néhányszor előfordult, hogy a súgó szerint követtem a lépéseket, mégis kaptam egy hibaüzenetet, amivel nem tudtam mit kezdeni.
Az előző két programhoz képest itt nem műveleteket végezhetünk, hanem elemzéseket. Nincs illesztés, nincs primer tervezés, van viszont R integráció, SNP feldolgozás és különböző összehasonlítások. Statisztikát kapunk mindenről, amiről csak akarunk.
Avadis NGS 1.3.1
Már a poszt megírása után tudtam meg, hogy létezik még egy program, ami érdemes lehet, hogy belevegyem az összehasonlításba. Ez a program nem más, mint az Avadis. A felhasználói felület és a program logikája teljesen olyan, mint a GeneSpringnek. Nem tudom, mi a kapcsolat a két cég között, de az biztos, hogy azonos kódbázisból dolgoznak.
A két program azonban teljesen más felhasználási területre készült. Amit hiányoltam a GeneSpringből, azt itt megkapom. Először is, lehet közönséges FASTQ fájlokat betölteni, akár tömörített formában is. A readek minőségéről kapunk statisztikát, mintha a FastQC-t használnánk. A BED állományt is minden gond nélkül be tudtam tölteni.
A readek illesztéséhez a BWA-ból ismert Burrows-Wheeler alapú illesztőt használ. Arról, hogy mennyire jó, talán majd egy másik bejegyzésben írok. Az illesztés minőségéről szintén képet kaphatunk néhány statisztika segítségével. Ez szintén hasznos funkció, és nem is láttuk másik alkalmazásban.
Szemben a GeneSpringgel, itt már tudunk VCF állományokat is importálni, valamint többféle variáció felderítő algoritmussal is kísérletezhetünk. A hagyományos SNP keresésen kívül felderíthetjük a strukturális változásokat, az SNP Effect Analysis segítségével pedig meghatározhatjuk, az adott variációnak milyen hatásai lehetnek. A különböző variációkat össze is hasonlíthatjuk, ezzel ez volt tesztünk egyetlen programja, ami mindegyik feladatot maradéktalanul végrehajtotta.
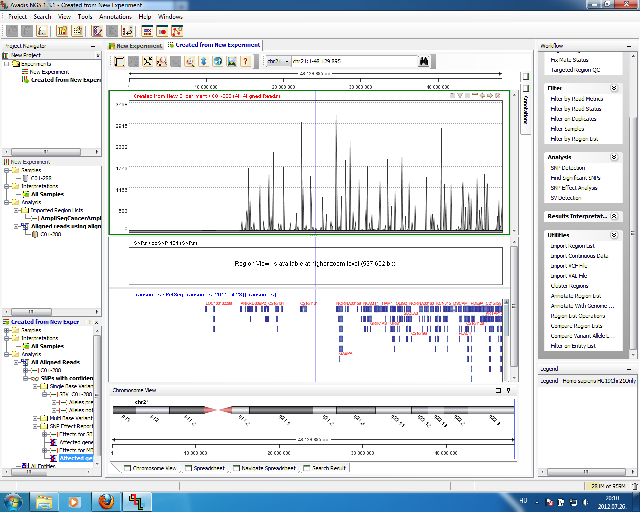
Lasergene Genomics Suite
A DNAStar programcsomagot régen használtam, méghozzá azt a verziót, amit minden magyar kutatóintézet használt. Egy PhD-s, aki fiatal korában egy külföldi cracker csapat tagja volt, feltörte a programot, és attól a pillanattól kezdve mindenhol azt használták. Már megjelent több újabb változata is a programnak, de itthon mindenki csak ezt a régi, de "ingyenes" verziót használta, mivel a benne található funkciók bőven kielégítették egy átlagos kutató igényeit.
Az új divathullámnak megfelelően van NGS alapokon nyugvó programcsomagjuk is, amit egyszerű halandó nem tölthet le, csak, ha regisztrál. A regisztrációt én nagyon komolyan szoktam venni, ezért olyan nevet szoktam választani, amiről lerí, hogy semmi köze a valóságoz. Az űrlap a végén arról tudósít, hogy egy munkanapon belül megkapom a letöltéshez szükséges információkat.
Sajnos ez nem igaz. Valóban kapok e-mailt. de arról, hogy válaszoljak olyan kérdésekre, mint például mennyi pénze van a cégnek, mi a kutatási területünk, stb. Itt kicsit felhúztam magam, ezért megírtam, hogy nem vagyok hajlandó egyetlen kérdésre sem válaszolni, amíg meg nem kapom a próbaverziót, valamint nem értem, hogy ez a bonyolult rendszer miért szolgálja az én érdekeimet. Válaszoltak, ahol leírták, hogy meg akarják védeni a programjukat az olyanoktól, mint én, akik 30 napig ingyen használják azt a napi munkájukhoz, de utána nem fizetnek. Megírtam, hogy ehhez a blogbejegyzéshez kellett volna a program, de mivel ez egy kicsi, magyar nyelvű blog, senkinek nem fog hiányozni a DNAStar terméke.
Partek
Ez az a program, amiről a poszt írásának idején értesültem először. Egy Google keresés adta ki eredményül. A próbaverzió megszerzése kisebb hadműveletet igényel. Először is egy webes űrlapot kell kitölteni, ahol a Hotmailt. GMailt és társait nem adhatjuk meg e-mail címként, csak céges e-mailt. Mivel még működik a régi egyetemi címem, azt adtam oda nekik. Kaptam egy levelet a következő munkanapon, ahol egy lmtools.exe letöltésére és futtatására szólítottak fel. Ez a gépről egy csomó adatot összeszed, becsomagolja, majd vissza kell küldeni a válasz e-mailben. Csak ezután kapjuk meg a licencet. Én egy hét után sem kaptam tőlük semmit, pedig megpróbáltam egy másik regisztrációt is új néven.
Összefoglalás
Még mindig nehéz dolga van annak, aki Windows alatt szeretne bioinformatikát művelni. Az Avadis NGS kivételével mindig akadt egy-két lépés, amikor el kellett volna hagyni a programot, hogy a feladatot máshol oldjuk meg. De sajnos ezek a programok nem arra vannak kihegyezve, hogy kooperáljanak más alkalmazásokkal, hanem ellenkezőleg, igyekeznek elszigetelni a felhasználót a külső világtól. Kevés az import/export funkció (bár meg kell jegyezni, hogy mind a Geneious, mind a CLC bővíthető pluginekkel). Érdekes kirándulás volt ez a klikkelős világban, de ha el akarom végezni a munkámat, akkor jobban megbízom az összehekkelt Bash scriptjeimben és a parancssoros programokban. De ha valakinek mégis egy összetett alkalmazás kell, akkor az Avadis megfelelő választás lehet.

