Néha a bioinformatikai munka olyan, mintha egy kísértet járta házat fedeznénk fel. Nappal. Tudjuk, hogy vannak szellemek, de azt hisszük, ezek csak éjszaka jönnek elő. A rontások tényleg nem szeretik a fényt, de nappal is vannak árnyékos helyek...
Nemrég gomba szekvenciákat kellett összeraknom. Pontosan nem tudtuk, milyen gombákból származtak ezek, ezért az ITS régió azonosítása nagyon fontos volt. Kétféle szekvencia állt rendelkezésünkre. Az egyik a szokásos Illumina volt, a másik Nanopore. Az Illumina mintavételénél a laborosok biztosak voltak benne, hogy Staphylococcus fertőzés is történt, ezért azokat különösen óvatosan kellett kezelni. A Nanopore-al ilyen probléma nem volt.
Több összeszerelést is végrehajtottam. Csak Illumina readek alapján, csak Nanopore szekvenciákkal és persze hibrid összeszerelést is végeztem. Nem akartam a fertőzéssel sokat bajlódni, ezért először csak a Nanopore szekvenciákkal épített genomot vizsgáltam. Az ITS régiók kópia száma 37 volt. Úgy gondoltam, ez nagyon sok, mert a referencia Saccharomyces genomokban 2 szokott lenni. Igazából volt más probléma is ezzel a genom összerakással (például a teljes mitokondriumnak csak 1/8-ad részét sikerül összerakni, az indelek száma meglepően magas volt), ezért tovább kellett lépni.
Megnéztem a hibrid összeszerelést is. Ez már ígéretesnek tűnt. Megvolt a teljes mitokondrium, viszont egészen biztos, hogy vannak benne olyan szekvenciák, amelyek nem gombából származnak, hála a fertőzésnek. Az összeszerelt scaffoldokat szűrni kellett valahogy. Először csak a baktérium genomokra illesztettem, majd ami arra felment, eltávolítottam. Ezután kerestem csak az ITS régiókat. Egy felet találtam! Ez elég szokatlan szám volt a 37 kópia után, amit a másik módszerrel határoztam meg. Megnéztem a szűrés előtti szekvenciákat, és két darab jött fel, akár csak a nagy könyvben.
Tehát túl szigorúan szűrtem. Nézzünk egy jobb szűrési módszert! Illesztettem a baktériumokra és a Saccharomycesre is. A két illesztésre adott pontszámot (bennfenteseknek: a Blast bitscore-t) pedig ábrázoltam egy koordináta rendszerben. A vízszintes tengely a baktérium illesztések pontszáma, a függőleges a gomba illesztés pontszáma. A szekvenciák egy része szépen szegregálódik. Ami a vízszintes tengelyhez van közel, az baci, ami a függőleges tengelyhez van közel, az gomba. Egyértelmű.
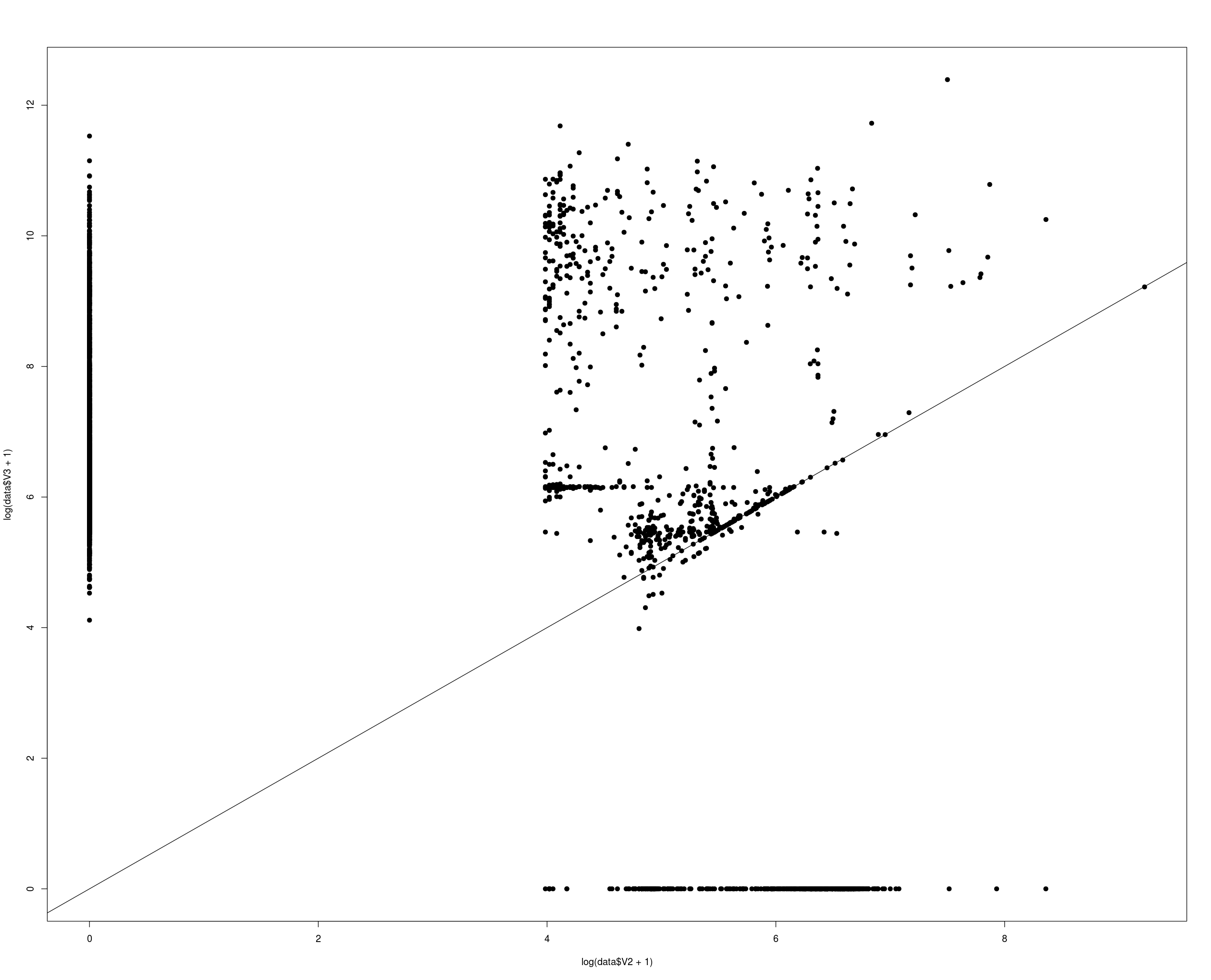
De mi a fene az a sok szekvencia az átló mentén? És miért vannak pontok az átlón? Hogyan lehetséges, hogy egy szekvencia közel olyan jól illeszkedjen a gombához is, mint a baktériumhoz? A horizontális géntranszfert sok mindenre rá lehet húzni, de erre nem.
Elkezdtem megnézni, miféle baci lehet ez. Az adatlapja szerint egy szekvenálásból származó szekvencia darabka. Egy hasonló kísérletből származik, mint amilyen az enyém is. Nekem is darabkáim vannak, nekik is darabkáik voltak. Én gombát akartam összerakni, ők baktériumot. Rá is engedtek mindenféle programot, ami talált géneket, érdekes régiókat, stb. Az NCBI pedig beemelte a referencia szekvenciák közé, és a reprezentatív baktérium Blast adatbázis része lett.
Ha viszont megnézzük, mivel mutat homológiát (bárki megteheti, van egy Run BLAST gomb jobb oldalon), akkor egy gombát találunk! Húúú, a szellem kibújt! Mégis csak elég hasonló a kísérlet az enyémhez! Amig nekem baci fertőzésem volt, addig nekik gomba jutott. De még én mindent elkövettem, hogy az idegen szekvenciákat eltávolítsam, addig ők ezzel nem sokat törődtek. Szinte belegondolni is rossz, hány ember használ ehhez hasonló adatokat, abban a hiszemben, hogy amit a szekvenciák leírásban találnak, az hiteles információ.
Természetesen az egyes adatbázisokban előforduló hibák nem újkeletű dolgok. Emlékszem, annak idején a MisPredre, ami az EnsEMBL hibásan detektált fehérjéit igyekezett kijavítani. Még én is "dinoszaurusz" DNS Blasztolását adtam fel egy állásinterjún feladatnak. De még a humán szekvenciák is okozhatnak szennnyezést a baktérium szekvenciákban.
Tehát mindenki legyen éber, mert bármikor beterítheti egy kis nyálka egy forduló után.
