Az emberi agy két dimezióval egész jól elboldogul. Három sem okoz túl nagy gondot. Adat elemzés közben viszont nem ritka, hogy sokkal magasabb dimenziószámmal kell dolgoznunk. Mivel az agyunkat nem tudjuk felfejleszteni a feladathoz, az adatokat butítjuk le olyan szintre, hogy mi is megértsük. Ennek több módja is van, ezek közül nézünk meg néhányat.
PCA
A régi jó főkomponens analízis segítségével könnyedén tudjuk redukálni a dimenziókat. Az adatok közötti távolságot is arányosan forgatja el, ezért a kapott eredmények további elemzések alapját is képezhetik. Például klaszterezhetjük azokat k-közép klaszterezéssel. A PCA determinisztikus, tehát mindig ugyan azt az eredményt fogjuk kapni. Paraméterezése nincs, ezért elrontani sem lehet semmit. Hátránya, hogy a többdimenziós teret úgy forgatja el, hogy az első főkomponens mindig a legnagyobb varianciával rendelkezzen. Ez az esetek legnagyobb részénél nem probléma, de néha az érdeklődésünkre számot tartó főkomponens nem az első lesz, ezért célszerű lehet a többi főkomponens ellenőrzése is.
t-SNE
Viszonylag új módszer a t-SNE. Az egy sejtes szekvenálások előretörésével lett egyre népszerűbb a bioinformatikában. A PCA-val ellentétben nem lineáris módszer, ami azt jelenti, hogy a pontok egymáshoz viszonyított távolsága torzul, emiatt nem célszerű az eredményt további elemzésnek alávetni, inkább csak vizualizációra használni. A pontok hasonlósága ugyanis nem egy fizikai távolságot jelent, hanem egy valószínűséget, hogy a két pont jellemzői mennyire hasonlóak. A t-SNE sztohasztikus, és erősen függ a paraméterezéstől, ezért több futtatás is adhat eltérő eredményeket. Cserébe viszont az összetartozó pontok látványosabb klasztereket alkotnak.
UMAP
Az UMAP annyiban hasonlatos a t-SNE-hoz, hogy nem determinisztikus algoritmus, és nem lineáris. A megközelítés viszont más. Az adatokat egy Riemann-sokaságként kezeli, amiről annyit mondanék csak (anélkül, hogy túl sok hülyeséget hordanék össze), hogy olyan, mint egy összegyűrt papír. Egy összegyűrt papíron nehéz megmondani egy pont helyzetét, de ha kisimítjuk, akkor például a jobb sarkától egy egyszerű koordináta rendszerrel leírhatjuk azt. Nos, az UMAP megpróbálja kitalálni, milyen összegyűrt papír illik legjobban az adatainkra. Amint ez megvan, szépen kisimítja azt. Hasonló dolog történik akkor is, amikor a Földet térképpé képezzük. Az algoritmus matematikai alapja megköveteli, hogy a pontok egyenletesen töltsék ki a teret. Mivel ez nem biztos, hogy minden adatra érvényes, ezért a pontokból egy gráfot építenek, ahol a szomszédokat éllel köti össze.
Lássuk mindezt a gyakorlatban. Először is töltsük le a GDC oldaláról az agyi tumorok normalizált expresszióját. Ez három tumor típust foglal magába (egy gliómát, egy glioblasztómát, és egy B-sejtes liómát. Ez utóbbiból csak kettő van). Az expressziós adatokat egy mátrixba kell gyúrni, amit a paste Bash parancs megtesz nekünk. Szükségünk lesz még egy metaadat táblázatra is, ahol a fájlnevek és a tumor típus található. Ezt a két táblázatot kell betöltenünk R-be.
library(umap)
library(Rtsne)
cancer <- read.table("matrix.tsv", header = T, check.names = F)
pcares <- prcomp(t(cancer))
umapres <- umap(t(cancer))
tsneres <- Rtsne(t(cancer))
metadata <- read.table("metadata.tsv", check.names = F, header = T, sep = "\t")
metadata$PC1 <- pcares$x[metadata$Sample,1]
metadata$PC2 <- pcares$x[metadata$Sample,2]
metadata$UMAP1 <- as.numeric(umapres$layout[metadata$Sample,1])
metadata$UMAP2 <- as.numeric(umapres$layout[metadata$Sample,2])
tsneres2 <- data.frame(Sample = colnames(cancer), X = tsneres$Y[,1], Y = tsneres$Y[,2])
rownames(tsneres2) <- tsneres2$Sample
metadata$TSNE1 <- tsneres2[metadata$Sample,2]
metadata$TSNE2 <- tsneres2[metadata$Sample,3]
A főkomponens analízis alapból a rendelkezésünkre áll a prcomp függvényen keresztül. Az umap és a t-SNE használatához külső csomagokra van szükség, ezeket töltjük be a library segítségével. Mindhárom függvény meghívása alapértelmezett paraméterekkel, megegyezik.
Ezután készítek egy eredmény táblázatot, ahol az egyes oszlopok a különböző módszerekkel előállított pontok koordinátái. A PC1 például a főkomponens analízissel elkészített eredmény x koordinátája. A táblázat sorai pedig az egyes tumor minták.
A koordináták sorrendje nem minden esetben egyezik meg a beadott minták sorrendjével, de szerencsére a táblázat sorainak nevei segítenek eligazodni. Ez alól a t-SNE eredménye a kivétel, ahol gyakorlatilag semmilyen információ nincs, hogy miként kell értelmezni az eredményeket. Feltételeztem, hogy a sorrend nem változik a bemeneti adatokhoz képest, de erre nincs semmi bizonyítékom. Ezért voltam kénytelen bevezetni a tsneres2 változót.
Lássuk az eredményeket:
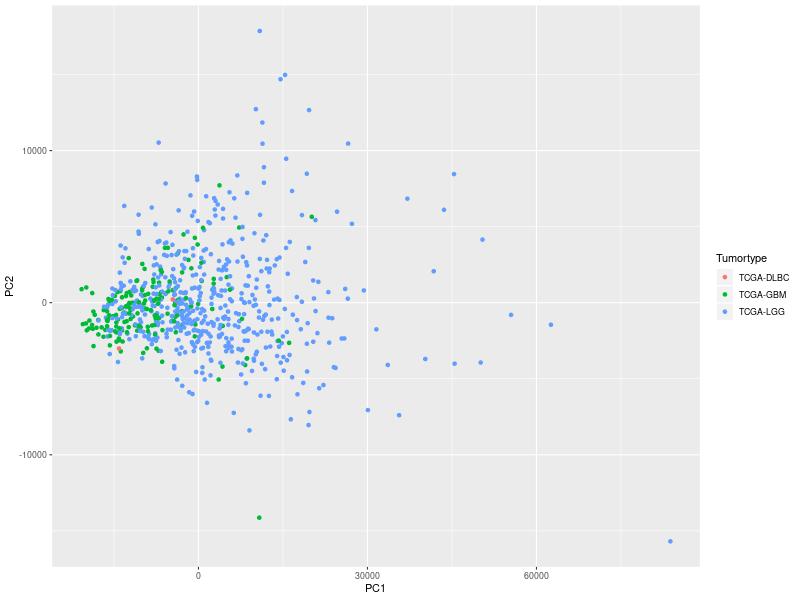
Főkomponens analízisnél a távolságok arányosak, ezért a kiugró értékek itt is elütnet a többi adattól. A glióma és blioblasztóma minták teljesen összekeverednek, ebből a nézetből nem látni semmilyen elkülönülést.
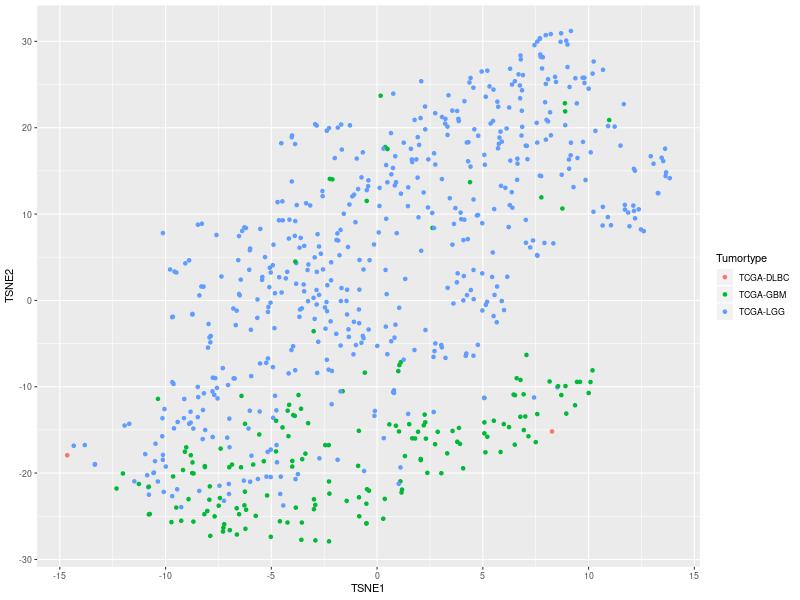
Csupán alapértelmezett beállításokkal dolgoztunk, de a t-SNE máris látványosabb eredményt adott. A tumor minták elkülönülése sokkal egyértelműbb. Látható, hogy glioblasztóma minták keverednek a kék pontok közé. Tudományos igényű elemzésnél célszerű lehet ezen adatok tüzetesebb vizsgálata, hogy megtudjuk, miért helyezkednek el messzebb a többi mintától. Az sem kizárt, hogy az adatokhoz jobban passzoló paraméterezéssel pontosabb eredményt kapunk.
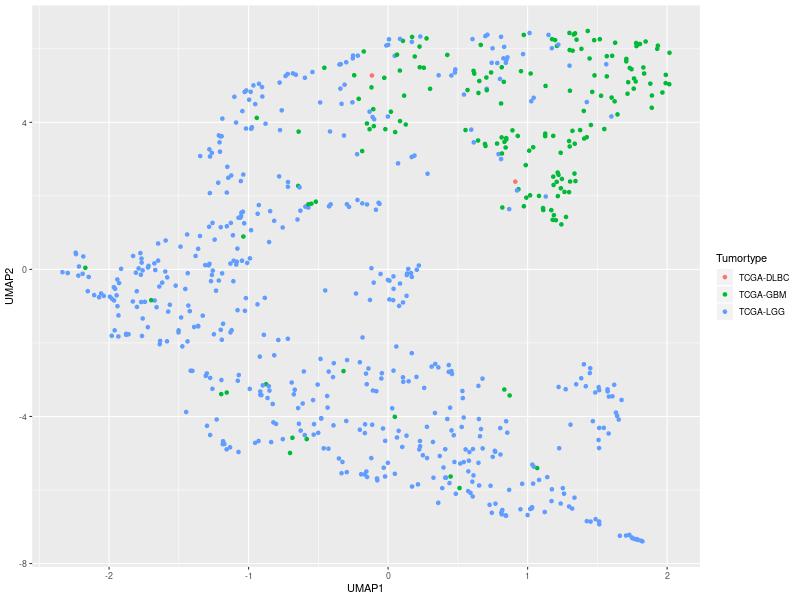
Az UMAP is hasonló eredményt ad, de a kép sokkal elnyújtottabb, az eltérő matematikai megközelítés hatására.