Itt az ideje, hogy kicsit jobban nekiugorjunk és létrehozzuk első saját bővítményünket. Először egy kis elméleti tudnivaló következik, majd kódolhatunk is.
Könyvtárszerkezet
A bővítményünknek a Cinema 4D plugins könyvtárában kell lennie egy új alkönyvtárban. Itt fog majd elhelyezkedni a lefordított bővítmény. Rajta kívül egy res könyvtárra van még szükség, ami nevéből következően az erőforrásokat tartalmazza. Mik is ezek az erőforrások? Ikonok, többnyelvű szövegek, dialógus ablakok. Az ikonok, mint ahogy a példa kódnál is megfigyelhető, egyszerűen a res könyvtárban vannak. A fejléc állományok a descriptions alkönyvtárban vannak. A dialógus ablakok megjelenítéséhez a dialogs könyvtárba kell elhelyezni az erőforrásokat. A többnyelvű szövegek a strings_nyelv-ben kapnak helyet, ahol a nyelv a következő lehet: us, de, jp, fr, it, es.
Bővítmény típusok
A Cinema 4D bővítményei hurok típusúak, ami annyit tesz, hogy a kódunk a program futásának bizonyos eseményei során futnak le. Ilyen esemény lehet egy menüelem kiválasztása, egy jelenet renderelése vagy az exportálás. Lássuk, mik lehetnek ezek:
- Parancs plugin: Egy menüelem kiválasztásakor aktiválódik.
- Jelenet export: Új formátumba menthetjük a jelenetet
- Jelenet import: Eddig ismeretlen fájlt építhetünk a jelenetbe
- Kép export: Bitkép mentése
- Két import: Bitkép betöltő
- Tulajdonság plugin: Az objektumok új tulajdonságot kaphatnak segítségével
- Eszköz plugin: Új eszközt adthatunk a Cinema 4D-hez
- Modellező plugin: Új objektum módosító eszközt hozhatunk létre vele
- Animációs plugin: Egyedi idősávot hozhatunk létre
- Shader plugin: A megjelenítést módosíthatjuk vele
- Jelenet plugin: A jelenet újrarajzolásánál aktiválódik
- Video render plugin: A renderelésnél hozhatunk létre különböző effekteket
- Üzenet plugin: Más bővítmények üzeneteit dolgozhatjuk fel vele
- Csomópont plugin: Új csomópontot vehetünk fel vele
- Egyedi GUI elem: Új grafikus elemet hozhatunk létre
- Egyedi adattípus: A beépített adattípusokat egészíthetjük ki
Regisztráció
Minden bővítmény egy egyedi számmal rendelkezik. Egyedi számunkat a Maxontól kell igényelni. Az itt bemutatott bővítmények csak egy hasraütött számot tartalmaznak, tehát előfordulhat, hogy már regisztrálva vannak. Ha nem használunk más bővítményeket, akkor nem kell aggódnunk, hogy esetleg konfliktusba kerülünk más bővítményekkel, de ha az a tervünk, hogy kereskedelmi forgalomba értékesítjük, mindenképp egyedi azonosítóra lesz szükségünk.
Forráskód
Bizonyára ez a rész érdekel mindenkit. Minden bővítmény három függvényt tartalmaz: PluginStart, PluginEnd, PluginMessage.
#include "c4d.h"
Bool RegisterCGMenu();
Bool PluginStart(void){
if(RegisterCGMenu() == FALSE) return FALSE;
return TRUE;
}
void PluginEnd(){
}
Bool PluginMessage(LONG id, void *data){
return FALSE;
}
A regisztrációt a PluginStart függvényben végezzük. Ha a regisztrációs sikeres, a bővítmény működni fog, ellenkező esetben kilép.
#include "c4d.h"
class CGMenu : public CommandData{
public:
virtual Bool Execute(BaseDocument *doc);
};
Bool CGMenu::Execute(BaseDocument *doc){
MessageDialog("My First Plugin");
return true;
}
Bool RegisterCGMenu(){
return RegisterCommandPlugin(1001999,"Hello World",0,AutoBitmap("cg.tiff"),String("hello"),gNew CGMenu);
}
Ez egy parancs plugin, amit a szülő osztály típusából állapíthatunk meg (CommandData). Egyetlen virtuális metódust tartalmaz, a parancs végrehatjását. Jelen pillanatban csupán egy üzenet ablakot jelenítünk meg egy egyszerű szöveggel. Végezetül a regisztráció mechanizmusát látjuk. Megadjuk az azonosítót, a menüben látható nevet, a bővítmény néhány tulajdonságát, az ikont, a tooltip nevet, legvégül az osztályt, amit regisztrálni kívánunk.
Ha elkészítettük első bővítményünket, kísérletezzünk bátran a paraméterekkel. Nézzük meg a dokumentációban, mit jelentenek, majd tapasztaljuk ki hatásukat.

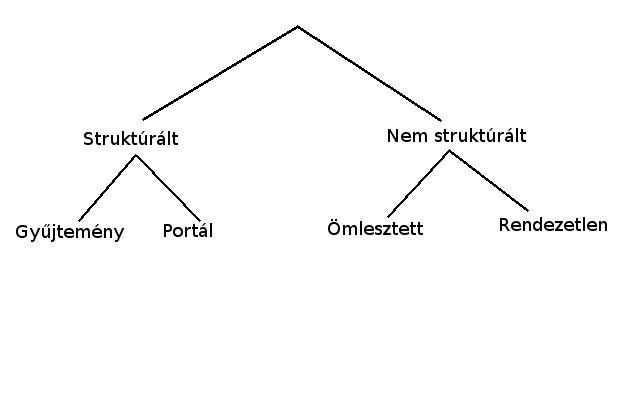 Nem struktúrált adatbázisok
Nem struktúrált adatbázisok