A cél
Már Function idején elkezdtem írni egy kisebb release-t, amolyan B-tervnek arra az esetre, ha Grass mégsem készülne el a Cubes-al. Szerencsére az erősen hiányos alkotást nem kellett bemutatni. Ott hallottam először az idei Experience-ről, ahol nem csak demókat nézhetünk, de még egy kisebb compot is szerveznek. Gondoltam ez a kis szösszenet megfelelő lesz rá.
A terv
A zene kiválasztásánál eleve arra törekedtem, hogy minél gyorsabban, minél kevesebb időráfordítással készülhessen el az alkotás, ezért a legrövidebb zenét választottam ki. Miután meghallgattam néhányszor, világossá vált, hogy három nagy egységből áll. Az első egy lágy dallam, ezt követi egy pörgősebb rész, végül visszatér az első téma. Tehát a három rész gyakorlatilag kettő, egy lassabb, és egy gyorsabb. Ez alapján készítettem egy részecske rendszert, meg egy alakváltó kockát. Írtam néhány shadert, hogy ne nézzen ki annyira ocsmányul, majd félretettem. Közben jött Topy, elmúlt a Function. Grassnak megmutattam a demó vázát, ő pedig ellátott hasznos tanácsokkal. A legfontosabb tanácsa, hogy be kell teríteni a képernyőt tartalommal. Az első verziónál a képernyő közepén ott voltak az alakzatok és csak ott mozogtak. Ezután már rengeteg alakzat mozgott minden irányba. Tényleg jobban nézett ki.
Fejlesztés
Nem akartam a már meglévő keret rendszerrel dolgozni, szerettem volna valamit újítani. Mivel a tartalom elég egyszerű, igyekeztem a kódba belevinni valami olyat, amit még nem csináltam. Például egy kis post processinget. Ennek megfelelően ez a demó már két lépésben állítja elő a tartalmat. Az első lépésben a testek és azok színe és mélységi információi bekerülnek egy framebufferbe. Ez a framebuffer több textúrában tartalmazza az információt. Később még ki lehet egészíteni normálokkal, rücskökkel, ambient occlusionnal, stb. Jelenleg csak a mélységet és a színeket tárolom. Miután feltöltöttem a framebuffert, jön a második lépés. Rajzolok egy, az egész képernyőt betöltő négyzetet, majd kirajzolom rá a textúrákat. A shaderek segítségével pedig különböző post processing effekteket rakok rá.
Igyekeztem jövő álló megoldást készíteni, de így is átesett két refaktoráláson, mert időközben rá kellett jönnöm, hogy az elsődleges tervek nagyon merevek. Még most is látok benne néhány limitációt, de már használható.
A használata így néz ki:
renderer = createRenderer(2, 2, width, height, aspect_ration);
Az inicializáló részben készítünk egy renderer változót, beállítjuk hány textúrába akarjuk elhelyezni a tartalmat (a mélységi információkon felül), mennyi shadert akarunk használni, valamint a textúrák szélességét és magasságát. (Az utolsó paraméterrel az oldalarányt állítjuk be, arról később lesz szó)
A rajzoló ciklusban a következő elemek találhatóak:
rendererFirstStage();
/* geometria kirajzolása, paraméter állítgatás */
rendererSecondStage();
/* post processing, shader paraméret állítgatás */
drawQuad();
Végül az erőforrások felszabadítása:
freeRenderer();
Ami nekem nem tetszik, hogy két helyen is kell paramétereket állítgatni. Sokkal jobb lenne, ha csak az első menet után lehetne mindent beállítani. Ezen még dolgozni fogok, de az eredmény már most magáért beszél! Az utómunkával jelentősen javult a látványvilág, már-már megközelíti egy igényesebb csapat gyengébb release-ét.
A másik újítás az ütem számláló, illetve annak egy kreatív elkészítése. Észrevettem, hogy a programozóknak sok manuális munkába kerül az ütemekre szinkronizálás, ezért leírom, hogy miként oldottam meg:
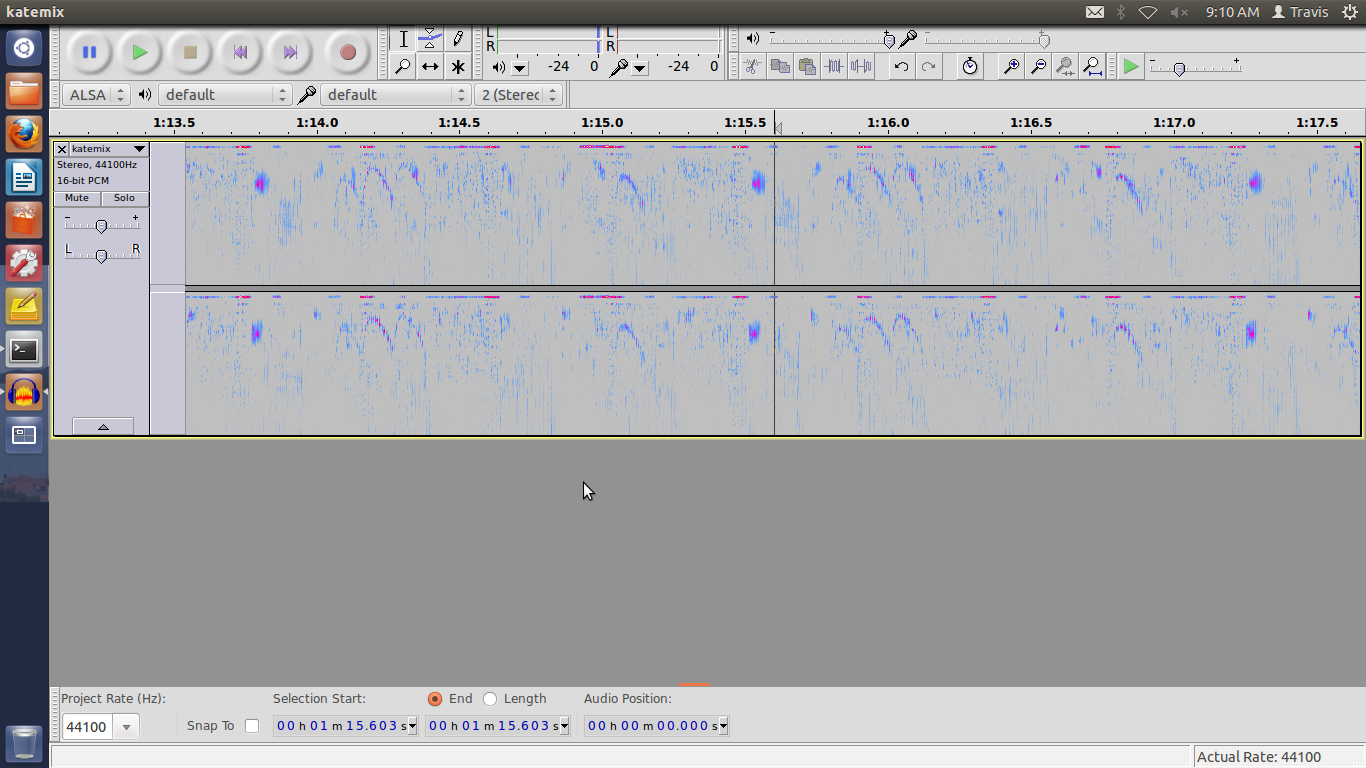
A zenét meg kell nyitni egy zeneszerkesztővel. Én az Audacityt használtam, mert azt ismerem, de biztos vagyok benne, hogy más programmal is meg lehet oldani. A hullámforma nézetet át kell állítani Pitch (EAC) módba, amitől az ismétlődő struktúrák szemmel könnyebben felismerhetőek (1. ábra). Ezután beazonosítjuk az ütemhez tartozó mintázatot, és feljegyezzük minden minta időpontját. A módszer pontossága függ a feljegyzett időpontok számától, de nem szükséges az összes időpont feljegyzése. A demó készítésekor én 10-12 időpontot jegyeztem fel. Egy táblázatkezelő vagy statisztikai program segítségével egy lineáris modellt illesztük a pontokra. Az egyenes egyenlete segítségével a demónkban kiszámolhatjuk az ütemek számát. A lineáris modell miatt a módszer hibatűrő. Ha egy-két pontnál rosszul jegyeztük fel az időpontot, az ütemszámláló attól még lehet pontos. Lássuk hogyan nézett ki nálam ez a gyakorlatban:
Feljegyeztem az időpontokat: 72.053 73.792 75.531 77.270 79.009 80.748 82.487 84.226 85.965 87.704 89.443 91.182
R-ben az egyenes egyenletének kiszámítása a következő módon néz ki:
> num <- c(1:12)
> time <- c(72.053,73.792,75.531,77.270,79.009,80.748,82.487,84.226,85.965,87.704,89.443,91.182)
> line <- lm(time ~ num)
> line
Call:
lm(formula = time ~ num)
Coefficients:
(Intercept) num
70.314 1.739
A num az egyenes meredeksége, az Intercept az X tengely metszéspontja (matematikailag: num*X + Intercept). Ha ellenőrizni akarjuk, hogy mennyire jó a módszer:
> plot(time ~ num)
> abline(line)
A demóba a következő módon lehet beépíteni:
int beatnum;
beatnum = (time - 70.314) / 1.739;
Problémák:
A demó készítése közben a Gimp egy új tulajdonságát is felfedeztem. Grass adott egy png formátumú grafikát. Gimppel megnyitva láttam, hogy Adobe palettával készült, de a program felajánlotta, hogy konvertálja RGB-be. Konvertálás után minden programban jól jelent meg, kivéve a demóban. Ott ugyanis el volt csúszva jelent meg a kép hasonlóan ahhoz, amikor 3 bájtonként akarjuk beolvasni a szín információkat, de a kép kevesebb bájton tárolja azt. Megpróbáltam mindent. Átkonvertáltam jpg-be, majd ismét png-be, minden kombinációban kapcsolgattam az exportáló funkciókat, de a kép csak nem akart megjavulni. Végül az segített, hogy kivágtam a teljes képet, majd beillesztettem új képként és azt elmentettem.
A másik álmatlan éjszakákat okozó hiba egy gömb volt. Nem akartam, hogy a demó háttere homogén legyen, ezért egy gömbbe helyeztem a jeleneteket. A gömböt Blenderben készítettem el, mert lusta voltam procedurálisan előállítani. A normál vektorokat felhasználva színeztem, de a "déli félteke" rendszeresen fekete volt. Sokáig nem is foglalkoztam a dologgal, csak bántotta a szememet. Először azt hittem, a shader kóddal van valami hiba, de miután nem találtam semmi furcsát és az internet szerint is jól számoltam, ismét betöltöttem Blenderbe a gömböt és megjelenítettem a normál vektorokat. A gömb felén a normál vektorok összes-vissza álltak. Két klikkeléssel megoldottam.
Ez az első demónk, ami 16:9-es oldalarányra optimalizált. Mivel nekem az összes monitorom a jó öreg 4:3-os aránnyal rendelkezik, így olyan megoldást akartam, ami minden képernyőn megtartja az oldalarányokat:
float aspect = 16.0 / 9.0;
float newheight;
newheight = (float)width / aspect;
glViewport(0, (height - newheight)/2, width, newheight);
Csakhogy a képernyő alján és tetején megjelenő fekete sáv mérete nekem nagyobbnak tűnt, mint amit megszoktam más szélessávú demóknál. A gyanúm akkor igazolódott be, amikor megjelenítettem a logókat, mert azok rajzoltak a fekete részre is. A hiba csak akkor jelentkezett, amikor nem 16:9 volt az oldalarány. Az a gyanúm, hogy a framebuffer mérete nagyobb, mint amit a glViewport-ban beállítok. Amikor a második lépésben kirajzolom a textúrát, ott már a glViewport és az ablakkeret között fekete részhez hozzáadódik a framebufferben eltárolt fekete rész is, amitől az oldalarány furcsának tűnik. A gyanúmat a logók kirajzolása is alátámasztja. A probléma az volt, hogy elfelejtettem, hogy az első lépésnek más nézet kell, mint a másodiknak. A gyakorlatban egy úgy néz ki, hogy két glViewport kell. A fenti megoldás csak a második lépésnél kell. Az elsőnél a következőre van szükség:
glViewport(0, 0, width, newheight);
Ezt beszúrva a renderFirstStage() függvénybe megoldódott a problémám.