Ez a demónk egy kísérletből született. Egy ideje nagyon foglalkoztat a házi motion capture gondolata, ezért elkezdtem tanulni az OpenCV-t (amiről hamarosan írok egy poszt-sorozatot). Úgy láttam, ez nagyon jó demo-alap lenne, Grass-al ezért elkezdtük tervezni a demót.
A másik lökést az adta, hogy tavaly megemlítettem egy AmiCon-on is, mivel foglalkozom, és Spenot felajánlotta, hogy kölcsönadja a Kinectjét, hogy azzal is kísérletezzem. Decemberben került sor az átadásra, egy hétig játszhattam vele. Gyorsan lementettem néhány videót, amit majd remélhetőleg fel fogok tudni használni a demóban.
Az egyetlen probléma az volt, hogy megközelítőleg 2GB méretűek voltak a mélységi információk. Ez még a legengedékenyebb compó-szabályzatnak sem felelnek meg. Szükség volt valami tömörítésre. Egy viszonylag egyszerű megközelítést alkalmaztam. Ahol a következő képkockában eltérés volt az előzőhöz képest, ott letároltam a koordinátát és a megváltozott értéket. Ezzel 7 és 11 MB méretűek lettek a fájlok. Úgy gondoltam további trükközésre nincs szükség.
Januárban videókat készítettem egy mobiltelefonnal, amit egy Amigás winchester beépítő kerettel rászereltem egy fotóállványra. A telefon videorögzítő képességével nem voltam megelégedve, mert gyakran minden figyelmeztetés nélkül leállt a felvétel. A másik probléma a színhűséggel volt. A fakó színek miatt a videofájlok feldolgozása volt problémás. Közben folyamatosan fejlesztettem egy kontúr detektáló programot, ami OpenCV-n alapult.
Februárban Grass nekem adta az egyik fényképezőgépét, ami már könnyedén ráült a fotóállványra, a videofelvevő képességével is elégedett lehettem. Egyedül az alacsony akkuidő hátráltatott. A kontúrokat már jól felismerte a program, el is készültek a demó felvételei. Annyit azért meg kell jegyeznem, hogy igyekeztem megkönnyíteni a program dolgát, amennyire csak lehetett, ezért fekete szabadidő ruhában, kesztyűben ugráltam a kamera előtt. Ez télen még nem volt akkora probléma (bár a hó a hátamon megolvadt), tavasszal viszont izzasztó volt.
Úgy emlékszem, ez idő tájt született meg a demo címe is, amit Grass talált ki, egy régi együttes után, akiknek hasonló nevük volt, legismertebb daluk pedig megegyezett az együttes nevével.
Márciusban a felvételekkel párhuzamosan írtam a demót, Grass pedig ellátott különböző tartalmakkal és ötletekkel a színekre vonatkozóan. Mivel elég messze lakunk egymástól, és nem volt demo kompatibilis laptopom, a gépemet megosztottam VNC-vel, így igaz, hang nélkül, de Grass is láthatta, hogyan alakul a produkció. Néhány esetben még az is ment, hogy az ötleteit azon nyomban megvalósítottam.
Néhány jelenetet leszámítva elkészült Revisionig, ahol mindenki tudja, mi történt. Parti után egy ideig nem csináltam semmit, majd ismét nekiláttam foltozni a hiányzó részeket.
Egyetlen nagyon komoly hiányossága volt/van a demónak, mégpedig a trianguláció pontatlansága. A demó ugyanis csak a körvonalat tölti be, majd felosztja háromszögekre és textúrázza a poligont. Sajnos egyes esetekben ez a lépés megszakad és üres lyukak maradnak. Rengeteg időt töltöttem, hogy megoldást találjak. Megpróbáltam egy automatikus módszerrel kiszedni a kereszteződött szakaszokat, de ezzel gyakran kivágtam a kontúr nagy részét, így inkább letettem róla. Próbáltam másik algoritmust készíteni, de azzal egy csomó időt elbíbelődtem, és nem készült el. A határidő viszont kérlelhetetlenül közeledett. Végül megpróbáltam csalni. A lyukakat valami gyors módszerrel befoltozom, de ezek általában konvexé alakították a poligonokat, ami ugyancsak rosszul nézett ki. A végén semmit sem változtattam meg.
A demóban igyekeztem OpenGL 4.1-t használni, mert így sok effektet shaderben tudtam programozni. Sokat segített, hogy nem kell újrafordítani a demót, csak a shader forrásában turkálva állíthatom a paramétereket. Ez főleg akkor volt a segítségemre, amikor a tömegközlekedésen kellett tuningolni a demón. Más racionális oka nem volt a választásnak. Egyébként is úgy szeretek demót írni, hogy valami újat tanuljak az elkészítése közben.
A produkciót mindenki megtekintheti a poue.net-en, YouTube-n. A forráskód elérhető GitHub-on. Ez utóbbinak az az előnye is megvan, hogy Gargaj elkészítette a Windowsos válozatát is. Ha lesz felvétel az élő közvetítésről róla, azt is belinkelem.




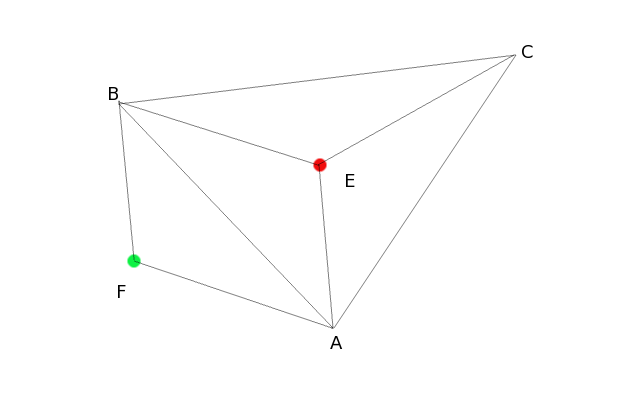 Ha megvizsgáljuk az AEB és az AFB háromszögeket, láthatjuk, hogy más a körüljárási irányuk. Az algoritmus tesztelésére készítettem egy C#-os programot, ahova idővel további triangulációs algoritmusokat is be fogok építeni.
Ha megvizsgáljuk az AEB és az AFB háromszögeket, láthatjuk, hogy más a körüljárási irányuk. Az algoritmus tesztelésére készítettem egy C#-os programot, ahova idővel további triangulációs algoritmusokat is be fogok építeni. Első versenyzőnk az
Első versenyzőnk az  A másik kereskedelmi termék a
A másik kereskedelmi termék a  Az első program, amivel átléptünk az ingyenes alkalmazások világába, a
Az első program, amivel átléptünk az ingyenes alkalmazások világába, a  Elérkeztünk a nézegető programok leghíresebbjéhez, az
Elérkeztünk a nézegető programok leghíresebbjéhez, az