Még tegnap este megnéztem kedvenc marketingesünk laptopját, mert állítólag elromlott. Az elromlott azt szokta jelenteni, hogy nem azt csinálja, amit a tulaj szeretne, ezért erős fenntartásokkal fogadtam a tüneteket. Végül sikerült megállapítani, hogy tele van a merevlemez hibás blokkokkal, ami lelassította a rendszerindítást. Egy éjszakán keresztül igyekezett a kis gép kijavítani a hibás szektorokat, de végül alul maradt.
Reggel a laptop tulaja úgy nézett ki a kávéja nélkül, mint egy zombi. Fel is ajánlottam neki egy szerepet még el nem készült poszt-apokaliptikus filmemben. De természetesen a legfontosabbak az előadások, így térjünk át ezekre.
A klinikai SNP keresés nagyban eltér a hagyományos kutatásban megismert módszerektől. Erről szólt az első előadás is. Az exome szekvenálás költséghatékony módja a variációk felderítésének, de felmerül a kérdés, vajon a szekvenálások mennyire befolyásolják a végeredményt? A vizsgálatok során a Complete Genomics és az Illumina szekvenálásokból kerestek variációkat. BWA-t, SOAP-Alignert használtak illesztésekhez és SOAPsnp-t, SOAPindelt, GATK-t, Samtoolst, SNPvert, GNUMAP-SNP-t SNPk és indelek azonosítására. A jó hír, hogy a variációk legtöbbjét mindegyik módszerrel meg lehet találni, de a gond azok a variációk, amelyek csak egyik vagy másik módszerrel találhatóak csak meg. A szekvenálás lefedettségének növelése sem megoldás, mert ezzel csökkennek ugyan a csak Illuminával megtalálható variációk (ez fals pozitívok jelenlétére utal), de a grafikonokból én azt láttam, hogy a Complete Genomics szekvenálásainál viszont növekedtek a variációk (ami viszont azt jelenti szerintem, hogy a valódi pozitívok száma alacsony). A részletek ebben a cikkben olvashatóak.
Ez után a depressziós előadás után egy kicsit vidámabb következett. Az előadó elmondta, hogy náluk azért működik az exome szekvenálás, az előzőekben hallott problémák ellenében. Erre hozott példákat is. Az egyik egy hibásan diagnosztizált egyén jó diagnosztizálása, a másik egy régi eset megoldása volt. Ennek ellenére nem tudja, hogy mennyire hatékony az exome szekvenálás, mert ők nagyon speciális eseteket dolgoztak fel. A norvég törvények nagyon szigorúak a humán klinikai minták biztonságával szemben, ezért náluk szóba sem jöhet semmilyen felhő alapú szolgáltatás. Előadás után megkérdzték tőle, hogy mi a véleménye a PacBio szekvenátorról. Azt válaszolta, hogy nagyon alacsony az áteresztő képessége, ami manapság már nem elegendő. A jövőre vonatkozóan annyit mondott, hogy kíváncsian várja a MinION USB-re csatlakoztatható szekvenálót.
Meghallgattam az IBM előadását is, így már képben vagyok, mire is van szüksége egy bioinformatikusnak. Az IBM hihetetlen mennyiségű számítási kapacitást és performanciát igyekszik a kutatók rendelkezésére bocsátani. Mit is jelent ez? 7,5 terabájt memóriát, Intel csúcsprocesszorokat (Sandy Bridge külön kihangsúlyozva). Ha valaki azt kifogásolná, hogy korunk bioinformatikai kihívásai I/O érzékenyek, azok megkapják a GPFS fájlrendszert. A ScaleMP már csak hab a tortán. Természetesen a bioinformatikust nem érdeklik a technológiai részletek, ezért a CLC-vel karöltve előre telepített vackok... izé bioinformatikus alkalmazások vannak. Személy szerint nem tetszett az erőforrások teljesen felesleges felhalmozása. 47 másodperc humán de-novo? Mire jó ez?
Hallgattam még egy klinikai sikertörténetet is, ahol az életkorfüggő fibropoliferatív repair genetikai okait keresték. Ez ugyanis az életkor előrehaladásával csökkenti egyes szervek működőképességét. Modellorganizmusnak transzgenikus egereket használtak, ahol genetikai targeteket igyekeztek keresni. Embereken észrevették, hogy a közös fenotípus mögött eltérő okok húzódnak. Egyes típust könnyebb azonosítani DNS típizálással, míg másokat mRNS mintázatuk alapján egyszerűbb meghatározni.
Egy másik klinikai diagnosztikai előadás arról beszélt, hogy ők még mindig Sangert használnak, mert náluk a cél, hogy minél kevesebb hibát ejtsenek a vizsgálatok során. Így is 10% körüli az emberi tényező okozta hiba. Ezt úgy igyekeznek csökkenteni, hogy két, egymástól független szakértő is elvégzi az elemzést. (Ezt egyébként a riportjukban is feltűntetik). A genetikai variációk feltételezett hatását a következő programokkal igyekeznek felderíteni: snps3d, sift, polyphen, pmut, panther. Természetesen mozdulni akarnak az újgenerációs szekvenálások irányába. Jelenleg egy CGC LIMS programot használnak, de KIWI néven egy új programot fejlesztenek GWT alapokon, Java EE szerverrel és MySQL liquibase adatbázissal. A forráskódot Mavennel rázzák gatyába.
Az utolsó klinikai diagnosztikával foglalkozó előadást azzal kezdték, hogy elég ritka, hogy egy genetikai eredetű betegség esetén, ha meg is szekvenálják a beteget, kiterjesztik a vizsgálatot más génekre, vagy rokonokra. XworX platformon fejlesztenek munkafolyamatot MutAid néven. Ez az eszköz inkább csak a funkcionális annotációban és a riport készítésében segít, mint a konkrét genetikai okok felderítésében.
Az IBM másik előadásán a hibrid architektúrájú felhőkről beszéltek. Valós időben akarnak elemzéseket végezni, vezeték nélküli nanoszenzorokból érkező adatokon. A beérkező adatokat pedig az örökkévalóságig tárolni. A hibrid architektúra azt jelenti, hogy vannak feldolgozás központú csomópontok, erős GPU-val, kis merevlemezzel, és I/O-ban erős csomópontok, de gyengébb processzorral. A csomópontok Hadoop-al, Oozival, Zookeeperrel vannak felhővé varázsolva.
A nap utolsó előtti előadása egy álomról szólt. Az EMIF projekt olyan új, hogy még weboldala sincs. Pontosan nem is értem mi lesz a lényege, de szeretnének egy platformot, ahol összegyűjtik a klinikai információkat és megosztják azokat, majd levonják a nagy következtetéseket.
A farmakogenomikáról szólt az utolsó előadás, illetve arról, hogy milyen eszközöket fejlesztettek, ami még farmakogenomikához is használható. Az egyik a FindBase, ami demoeffekt-szerűen vizualizálja a keresési eredményeket. Mutációs függőségi gráfokat is tartalmaz. A másik termék a Goldenhelix.
A kiállítok között feltűnt a Virtalis. Kiraktak egy 3D projektort, aktív szemüveggel egy PyMol segítségével megjelenítettek egy modellt és megpróbálták elmagyarázni, hogy ez a jövő. Azért, mert a filmeket már nem tudják eladni 3D nélkül, az nem jelenti azt, hogy ettől jobban fog menni a tudomány. Beszélgettem egy kémikussal, aki szintén a kiállítók között volt. Szerinte, például a fehérje szerkezeti vizsgálatnál teljesen felesleges a 3D vizualizálás, mert nem az a lényeg. Egyetértettem vele.
BioIT World 2. rész
2012.10.10. 23:32 Travis.CG
Szólj hozzá!
Címkék: cloud computing bioinformatika
BioIT World 1. rész
2012.10.09. 21:35 Travis.CG
A vonat sebesen szeli át az országot, hogy elérjen Bécsbe, a BioIT World konferencia színhelyére. A cég, amelynek alkalmazásában állok, itt akarja népszerűsíteni legújabb termékét, nekem meg sikerült kiharcolnom, hogy eljöhessek én is. Az eljövetel egyik feltétele az volt, hogy végighallgassak egy értékesítési fejtágítót, amit egy igazi "értékesítő zseni" tartott.
A modern időknek megfelelően az előadó és a hallgatók egyike sem tartózkodott 20km-nél közelebb egymáshoz, ezért ahogy közeledett az idő, egyre biztosabb voltam benne, hogy ez valami távoktatás jellegű dolog lesz. Habár szinte mindenki, aki nálunk dolgozik, Linuxot használ, nagyjából 5 perccel az előadás előtt derült ki, hogy GotoMeetingre lesz szüksége annak, aki meg akarja hallgatni azt. Mondanom sem kell, ez az a fajta Java alapú program, ami nem támogatja a Linuxot. Negyed óra szerencsétlenkedés után Skype-ra tértünk.
Az előadás abból állt, hogy az értékesítő zseni hangja percenként elhalkult, amit rögtön a marketingesünk ordítása követett: Nem hallak, nem hallak! Talán attól félt, hogy ő is elhalkul, ezért ordított. Ennél a pontnál kikapcsoltam a mikrofonomat, hogy ne hallják a nevetésemet.
Nem előadás az előadás, ha nincs prezentáció. Ezt e-mailen kaptuk meg, hogy nézzük, miközben hallgatjuk a marketinges kétségbeesett kapcsolat-fenntartó kiáltásait. Ez egy PowerPoint fájlt volt, amit egyedül mi, Linuxosok tudtunk megnyitni. Talán nem okozok nagy meglepetést azzal, hogy elárulom, a marketingesünk Windowst futtat. Habár korlátozott képességekkel a GMail is meg tudja nyitni ezt a fajta állományt, újabb tíz perc úszott el azzal, hogy DropBoxon keresztül újra elküldték neki a fájlt, amit csodák csodája, így sem tudott megnyitni. Hiába, a fájl megyithatósága nem az elküldés módjától függ.
Ezt leszámítva elég eseménytelen volt az előadás. A legfontosabb üzenet: kérdezzünk, hagyjuk a potenciális vevőt beszélni, tudjuk meg, mire van szüksége.
Mivel ez egy tudományos konferencia is, ezért vittünk egy nagyon tudományos posztert is. A többi bioinformatikussal beszélgetve számomra kiderült, hogy ezt a posztert egyetlen tudománnyal foglalkozó ember sem látta. De látták a menedzserek, marketingesek, igazgatók, sőt ők készítették. Remélem a konferencián legalább én is megnézhetem. Holott homályosan emlékszem, hogy voltam egy megbeszélésen, ahol ezt a posztert akartuk megvitatni. De csak addig jutottunk, hogy milyen betűtípussal legyen elkészítve a táblázat. Azért a nyers adatok feldogozásában benne volt a kezem.
Azért nem lehet okom panaszra. A csapatból nekem engedték meg egyedül, hogy meghallgassam az előadásokat. Igyekszem beszámolni az új trendekről, érdekes vizsgálatokról és minden egyébről, mi valamilyen okból felkeltette az érdeklődésemet.
Ennyi bevezető után lássuk, mit is tartogatott az első nap, ami valójában a második, mert az első napon nem voltam. Az előadások két szekcióban folytak. Az első a klinikai szekció volt, míg a második az IT témájú előadásokat foglalta magába.
Az első előadás az epigenetikus biomarkerekről szólt. Az epigenom változásai a hiszton modifikáción keresztül komplex betegségeket okozhatnak. Mindezt akár egészséges genotípus mellett is. Az epigenetikus regulátorok (pl. TET2, EZH2) mutációja különböző rákos megbetegedésekhez vezetnek, de hatásuk reverzibilisen visszafordítható különböző gyógyszeres kezelésekkel. Az epigenom vizsgálata az előadás tanúsága szerint olyan metasztatikus karcinómáknál is hasznos lehet, ahol az elsődleges gócpont nem ismert. Az előadás azt is taglalta, milyen szerepe lehet az epigenomnak a személyre szabott gyógyászatban.
Az OpenBEL megoldja a szervezeteken belüli tudás megszerzését, megosztását. Az előadást nem hallottam, csak az utolsó diát láttam meg, és leírtam a címüket.
A második előadás a nagy adat kihívásáról szólt, és miként fogjuk (vagy inkább unokáink fogják) megoldani a felmerülő problémákat. Egy rendszer működésének megértése és a belőle nyerhető információk feldolgozása két különböző folyamat. Ha egy rendszer bonyolult, nem feltétlenül ismételhetőek meg a hozzá kapcsolódó kísérletek. Az agy például folyam-szerű elven működik. Ha ezt akarjuk utánozni, akkor a hagyományos Turing-elven működő számítógépek helyett valami mást kell létrehozni. Nagyjából ennél a pontnál vesztettem el a fonalat. Ezután a kvantum fizikáról, kozmológiáról folyt a szó, majd az önmagukat másoló makormolekulákat egyszerűen univerzális Xerox gépekké redukálta az előadó. Mikor az egyik dián egy Naprendszer méretű holografikus memória térképe jelent meg, ami már most is jelen van (és erre a "bizonyíték" az autizmus, az epigenetikus öröklődés és a deja vu), akkor adtam fel a reményt, hogy ebből bármi értelmes lesz. Az előadást tovább rontotta, hogy valami angol-orosz-francia keverék nyelven szólt. Még egy maradandó "tudományos" információ is elhangzott: A hosszú életű emberek ősszel születnek.
De szerencsére értelmes előadás is volt. A pool-seq módszerről is hallgattam egy előadást. Ennek a célja, hogy egy populáció DNS-t ekvimoláris mennyiségben összekeverik, majd megszekvenálják. A felgydúsuló SNP-k nem egyén, hanem populáció szinten lesznek jellemzőek. A módszer hátránya, hogy a strukturális variációk és mappelési hibák nagyban megnehezítik a kiértékelést. Az így keletkezett adatokat a PoPoolation nevű szoftverrel érdemes feldolgozni. Az is elhangzott, hogy az SNP analízis után nem érdemes GO vizsgálatot végezni, mert a GO analízis microarray módszerekre lett kihegyezve, nem SNP analízisre.
Izlandon sem ülnek a babérjaikon a bioinformatikusok. Készítettek egy olyan klinikai szekvencia elemző programot, ahol a megszekvenált egyénről megállapítja, hogy hány százalék esélye van a különböző karcinómákra. A rendszer alapja egy GOR (genomic ordered relations) nevű rendszer. Ez egy Java nyelven implementált program, ami Unix-szerű csővezetékeket utánoz, mindezt egy klaszteres környezetben, tehát a lekérdezés és elemzés szinte valós időben folyik. A deCODEhealth nevét még biztosan fogjuk hallani.
A Panasas cég is bemutatta párhuzamos adattároló eszközét. Előnye, hogy egy doobzban redundánsan ott egy csomó tárhely és a hozzá tartozó blade szerverek. Van egy objective RAID architektúrájuk, ami dinamikusan változtatja a RAID konfigurációt, attól függően, hogy mennyi és milyen típusú adatom van. Természetesen van benne SSD is, a kis adatoknak. Mindez egy saját fejlesztésű pNFS fájlrendszeren keresztül érhető el.
Megismertem a DARIO nevű programot is. Ez a nem kódoló RNS-ek (ncRNA) osztályozását végzi. Egy tanulóalgoritmusnak beadták a különböző RNS-ek mappalési mintázatát, majd a program megállapítja egy BAM fájlból, hogy milyen típusú RNS-eket sikerült megszekvenálni.
Egy olasz prof elmagyarázta, hogyan lehet a nagy adatmennyiséget tudássá váltani. Rögtön úgy kezdte, hogy nem is ért a biológiához, de ha a folyamatokat formális állapotváltozásokra írta át, akkor már mindjárt tud vele dolgozni. Szerinte a nagy adatmennyiséget nem a flopok számának növelésével kell megoldani, hanem új programozási modellek bevezetésével. Ez alatt nem új programnyelveket és új könyvtárakat ért, mert ezek nem programozási modellek. Ennek ellenére ő és munkatársai készítettek egy C++-os könyvtárat, ami jó az összes felvetett problémára. Lehet, hogy még ő sem kész új programozási modellek kifejlesztésére?
A Sztakiból is volt egy előadó. Ők egy olyan felhő alapú megoldást mutattak be, ami elrejti a különböző felhő alapú szolgáltatások közötti különbségeket, sőt egy újabb absztrakciós réteggel még a munkafolyam programozást is egyszerűsítették. Ők három féle felhasználót különböztettek meg. Vannak, akik a sci-bus rendszert fejlesztik. Ez a legalsó réteg, ők fejlesztik a kapcsolódást a különböző felhő-alapú rendszerekhez. Vannak a munkafolyamat fejlesztők, akiket nem érdekel a felhő, csak az, hogy működjön a programjuk, végül vannak a végfelhasználók, akik csak a programot akarják paraméterezni, de nem érdekli őket az infrastruktúra. A rendszerük CloudeBrokert használ, amivel megvalósítja a különböző felhő rendszerek közötti átjárást és LifeRay-t a folyamatok programozásának egyszerűsítésére.
A poszterek közül a GENALICE ragadta meg a figyelmemet. Ez egy új illesztőprogram, ami természetesen gyorsabb és érzékenyebb, mint bármi más a Földön.
A kiállításunkat elég sokan megtekintették a kávészünetekben. Este hétig fogadtuk az embereket. Mikor már elkezdtek fogyni az emberek, a céges projektoron, ahol eddig a marketing filmünk ment végtelenítve, véletlenül két CG demó is lefutott, csak a miheztartás végett.
Szólj hozzá!
Címkék: cloud computing bioinformatika
Statisztika, az ördög műve
2012.10.06. 21:38 Travis.CG
Röviden prezentálom a HIV fertőzés emberekre gyakorolt hatását: Íme egy ember, aki HIV pozitív volt:
Íme egy ember, aki nem volt HIV pozitív:

Vannak, akik megfertőződnek, de nem alakul ki náluk a tünetegyüttes, és vannak olyanok, akiknél igen.
Ennyi a beszámoló. Az olvasó, biztos vagyok benne, hogy nem tudott meg többet a HIV fertőzésről, és bizonyára úgy érzi, hogy kihagyok valamit. Pedig egy ehhez hasonló bioinformatikai elemzést olvastam. A szerző összehasonlított két illesztőprogramot. Talált több, mint 11 ezer variációt, majd készített 4 képet az illesztésekről, ahol prezentálta, hogy egyik program illesztésében megtalálható egy bizonyos variáció, a másik illesztésben pedig nem.
Udvariasan arra kértem, hogy legyen szíves valami statisztikát mellékelni. Semmi bonyolultat nem akartam, csak pár százalékot, hogy egyik program mennyit talál meg, és a másik mennyit talál meg. Esetleg egy PPV-t számolhatna, hogy a megtalált variációk hány százaléka valódi, és mennyi fals pozitív.
Természetesen ilyet nem kaptam, mert a riportot készítő főnöke elmagyarázta nekem, hogy ezek a statisztikák csak arra jók, hogy elfedjék a lényeget, ezért nem szereti őket. Tehát a fenti példa "beszámolóm" teljes, mert nem fedi el a lényeget. De az olvasó mégsem tudja, hogy mennyire veszélyes ez a vírus. Tehát ez a főnök ezzel a kijelentéssel a hozzá nem értését bizonyította.
A kegyelemdöfést az adta meg, hogy a riport készítője is írt. Mint leírta, ő sem szereti a statisztikát, mert neki kell kiszámolnia. Tehát van egy dilettáns főnök és annak egy lusta beosztottja. Készítenek valamit, ami teljesen használhatatlan, és megmagyarázzák, hogy az minden igényt kielégít.
A statisztika rossz alkalmazása valóban elfedhet egyes jellemzőket. Ilyen például, mikor csak az átlagot prezentálják, a szórás nélkül. De a statisztika nem gonosz. Nem azért van, hogy megkeserítse az emberek életét, hanem azért, hogy felfedje az összefüggéseket. Egyedi esetek felsorolása nem megoldás. A másik, hogy a statisztikát nem szeretni vagy utálni kell. Szeretni lehet süteményt, filmet vagy háziállatokat. Metódusokat viszont használni kell, nem szeretni.
Szólj hozzá!
Címkék: filozofálás
Function2012 II. rész
2012.09.30. 02:35 Travis.CG
Második nap az előadásokra értem vissza. Főleg a játékipart megcélzó fejlesztésekről beszéltek. A People Can Fly nevű cégtől megtudhattam, mit is csinálnak egy első osztályú játékfejlesztő cégnél a programozók. Ez az a szint, amit személyesen soha nem fogok megtapasztalni. A második előadás a Havoc fizikai motorról szólt. Szárazabb volt az előzőeknél, számomra csak a fizikai modellezésnél kezdett érdekes lenni. A middleware fejlesztés létjogosultságát alátámasztó gazdasági kimutatások sajnos nem érdekeltek.
Isti, akit az Amiga Klubból ismertem, hatalmas release készítésben volt. Ez abból állt, hogy embereket keresett, akik demót írnak helyette. A plazmakockájának textúráját Charlie írta, a kocka kódját egy OpenGL példaalkalmazásból lopta. Egy kicsit segítettem neki én is, még egy hónappal a parti előtt (bár meg kell jegyeznem, sok hasznomat nem vette.) Itt Blala pakolta össze a különböző helyről származó kódokat, míg zenét Ferris adott, akit elvitt valami elektronikai boltba. Mondtam is Istinek, hogy demotoolt használ. Humán demotoolokat.
Segítséget kértem én is a profiktól. Egy ideje gondolkodom rajta, milyen design patternt érdemes használni, hogy ne kelljen foglalkozni az OpenGL kód és a shader kód közötti függőségekkel. Értem ez alatt, hogy a vertex bufferek, uniform változók nevei olyan függőségeket kényszerítenek a programozóra, ami miatt megnő a hibalehetőségek száma.
Azt a megoldást kaptam, hogy először a shadereket készítsem el egy eszköz segítségével, majd ehhez igazítsam a program kódját. Érdekes koncepció, én eddig fordítva csináltam. Volt egy stabil rendszer kód, és a shadereket heggesztettem, amíg nem lettek jó. Ez azért volt jó számomra, mert fordítás nélkül tudtam változtatni a demón.
A SidRip Alliance koncertjét a termen kívül élveztem, és nem voltam egyedül. Nem a zene mindősége üldözött ki, hanem a hangerő, amit a vén csontok fülei már nehezebben bírnak. De cserébe Ratmantól megtudtam, milyen napjai vannak manapság egy rendszergazdának. A beszélgetés megvilágította, hogy nem vagyok egyedül, amikor a hozzá nem értéssel kell harcolnom. Csak mások még rosszabb helyzetben vannak, mert őket éjszaka is zargathatják. Kedvenc történetem a következő volt:
- Este hívnak, hogy nem megy a szerver. Mikor megkérdeztem, mi a hibajelenség, a kagylót letették. Kiküldtem egy embert, nyomozza ki, mi a hiba. Egy óra múlva hívott, hogy a problémát elhárította. A nyomtató begyűrt egy papírt.
Ebből is látszik, vannak problémák.
A zenék között most nem volt olyan, ami mélyen megfogott volna. A grafikák között volt néhány igazán érdekes, de erről Grass majd részletesen ír a scene.hu-n. Amit nagyon vártam, az a wild demók, hiszen érdekeltek voltunk benne. Az első pár release a szokásos, "gyorsan dobjunk össze valamit" szellemében készült. Majd jöttek az igazi nagyágyúk. Meglepően jó releaseket láttam. Volt technikai jellegű, de igazán ötletes darabok is. Azt hiszem, a dobogóról lecsúsztunk.
A 256b intrók száma is magas volt. Itt is akadt olyan, amire érdemes emlékezni. A nagyobb méretkorlátú intrók már nem voltak annyira emlékezetesek, de a Dilemma nevű csapat rendesen kitett magáért, szerintem nyerni fognak. Platformok terén sem lehet ok panaszra. A PC-s indulók mellett volt Amiga is. A magyar Amiga scene (vagyis Charlie) ismét képviseltette magát.
Gargaj figyelmeztetett minket, hogy nagy számú demó várható. Nem is hazudott. Több, mint 10 induló volt. Egy részük lelkes scenerek örömmunkái voltak. Azonban voltak igazán figyelemre méltó alkotások is. A Brainstorm nem adta ki a demóját valami bug miatt. Tehát amit a legjobban vártam, elmaradt.
Az eredményhirdetésen kiderült, hogy 5. lett a Cubes.
Szólj hozzá!
Címkék: demoscene parti riport
Topy Function-re megy
2012.09.30. 02:23 Travis.CG
Topy, a német demoscene egyik nagy egyénisége, elmondása szerint igyekszik minél több partira eljutni. Eddig ez nem okozott neki különösebb problémát, de idén egy autóbaleset következtében eltörött a lába. Talán mások az ő helyében igyekeztek volna kíméletesebb életmódot élni, és inkább a gyógyulásra koncentrálni, ő mégis úgy döntött, a demopartikat nem fogja kihagyni.
El is jutott a WeCan nevű lengyel partira, és úgy döntött, ha már itt van Kelet-Európában, a Function-t is meglátogatja. Egyetlen akadály volt csupán, megközelítőleg 10 nap a két esemény között. A szállodák nem olcsóak, még a német pénztárcákhoz képest sem, ezért Topy a scene közösség segítségét kérte.
A fórumon leírta, hogy mi a problémája, mit szeretne és várta, hogy milyen ajánlatokat kap. Az átlagos scener 30 év körüli, tehát jó eséllyel van családja. Náluk egy törött lábú ember befogadása szóba sem jöhet. Gondoltam egyet, és írtam neki. Igaz, nem élek Budapesten, ami a közlekedést megnehezíti, de nem tartottam valószínűnek, hogy leküzdhetetlen akadályokkal kellene szembenézni.
Rajtam kívü egy ember tett még felajánlást, de később ő is visszakozott. Topynak nem volt más választása rajtam kívül. Bevallom, én is meginogtam, de úgy döntöttem, ha már egyszer tettem egy ígéretet, akkor nem változtatok rajta. Így történt, hogy szeptember 19-én a repülőtéren vártam, hogy megérkezzen Topy.
A kocsi, amivel közlekedett, nagyobb volt, mint gondoltam. A buszra alig tudtunk felszállni, de szerencsére segített a sofőr. Topy elmondta, hogy már nincs akkora szüksége a kocsira, de mégis kénytelen cipelni, nem hagyhatja ott valamelyik parti helyszínén. A lépcsőkkel sok esetben akadtak problémáink. Ez az utazás ahhoz is nagy segítséget nyújtott, hogy másik szemmel lássam a közlekedést kerekes székkel.
Majdnem három órát vett igénybe az út, de végül eljutottunk a lakásomig. Beszélgetésünk során kiderült, hogy dolgozott hangmérnökként, belekóstolt a filmgyártásba, ahol a világító, villogó, de valódi funkció nélküli elektromos ketyeréket készített (tudjátok: a piros vagy a kék drótot vágjam el?) és független játékokhoz készített zenéket. Ha viszont úgy hozza a sors, pólókat árul Atari, Amiga feliratokkal.
Elmesélte, hogy üzemeltet egy repaidcafe-t Kölnben. A kezdeményezés lényege, hogy csere alapon ki-ki megjavítja a másik elromlott, ősrégi eszközeit. Ha valaki visz oda egy egyközt, legközelebb neki is meg kell javítani valamit. Ezt én is egy szimpatikus hozzáállásnak tartom. Szerintem sem kell kidobni egy eszközt és újat venni, csak azért, mert egyszerűbb.
Amíg nálunk tartózkodott, bár nem sokszor mozdult ki a lakásból, (pontosabban még a laptopját sem hagyta el), mégis élénk társasági életet élt. Virtuálisan még a barátnőjével is randizott. Mindketten letöltötték ugyan azt a filmet, majd chaten megbeszélték, mit látnak.
Topy félig ír, ezért főzött nekünk egy igazi ír különlegességet. Sajnos a nevét nem tudom. Birkahús volt megsütve hagymával, párolt zöldségekkel. Leveles tésztával letakarta és úgy is megsütötte. A hús olyan volt ízre, mint a vadas.
Topy feladata a Function idején a SceneSat élő közvetítése volt. Tehát annak, hogy a világ más tájain hallani lehetett a Functionon elhangzottakat, egy kicsit nekem is köszömhető.
Szólj hozzá!
Címkék: demoscene
Function2012 I. rész
2012.09.29. 16:22 Travis.CG
Idén is eljött a Function. Pénteken Topyval elindultunk a parti helyszínére. Kerekes székkel és felszerelésekkel együtt ez egyáltalán nem egyszerű feladat. Felszállni a villamosra volt a legnehezebb feladat, mert középen ott volt az a hülye korlát. Rajtam volt a saját hátizsákom, Topy hátizsákja, jobb kezemben a ruhás táskája, a ballal pedig az összecsukott kerekes széket vittem. Ő mankóval ment.
Mikor megérkeztünk, mondtam, el sem hiszem, hogy itt vagyunk. Ő meglepetten rám nézett: Miért, nehéz volt idejönni?
Lepakoltunk, majd elkezdtünk beszélgetni az emberekkel. Mellém egy japán scener telepedett, aki rögtön ellátott Mangás nápolyival. A megnyitó nem volt túl ünnepélyes, de nem is voltunk olyan sokan, hogy fontos legyen.
Láttam Arcee játékát. Autóversenyt készített a Tricky Truck motorjával. Mu6k mtatott 5 másodpercet a készülő demójukból, ami el is vette a kedvemet attól, hogy beadjak bármit is. Ennek ellenére javítottam egy kicsit az egyik demómon. Velem szemben egy akkora laptop került, mint egy TV. A tulaj megnézte a szánalmas release csökevényemet, majd elképeve mondta, hogy ez fantasztikus, ő ilyet soha nem tudna csinálni. Akkor mire jó az a nagy laptop? A game compó elött hazamentem.
Szólj hozzá!
Címkék: demoscene parti riport
Előfeldolgozás OpenCV-vel. Második rész
2012.09.25. 21:51 Travis.CG
Nyers kép
Ha birtokunkban vannak a feldolgozandó képi információk, az első lépés, hogy ezt a programunk számára elérhetővé kell tenni. Három különböző módon olvashatunk be képeket:
Webkameráról a cvCaptureFromCAM, videóról a cvCaptureFromFile használható. Mindkét parancs egy CvCapture struktúrát ad vissza, ahonnan a cvQueryFrame segítségével kinyerhetjük az egyes képkockákat. Ha csak egyetlen képpel dolgozunk, akkor a cvLoadImage betölti nekünk azt.
Amennyiben egy köztes állapotot tárolására nekünk kell létrehozni egy képet, a cvCreateImage-t használhatjuk.
Bármelyik módszert is használjuk, eredményül egy IplImage-t kapunk. Ez a nyers képünk. Attól függően, hogy mi a célunk, a képet különböző előfeldolgozásnak kell alávetni. Sok parancs hatása hasonló, mintha Photoshop szűrőket használnánk, tehát gondolhatunk úgy is az OpenCV-re ezek alapján, mint egy programozható grafikus alkalmazásra.
Photoshop imitálás
Az egyik legfontosabb feladat, hogy különböző színterek között át tudjuk alakítani a kapott képet. Erre szolgál a cvCvtColor függvény. Három paramétere közül az első kettő a kép, amiről konvertálunk, a kép, amire konvertálunk. Az utolsó paraméterrel adjuk meg a konvertálás módját. A módokat konstansokkal adhatjuk meg. Ha szürkeárnyalatos képet akarunk kapni egy RGB színterű képből, a következő konstanst használhatjuk: CV_RGB2GRAY.
Szintén alapvető fontosságú feladat a képek átméretezése. A cvResize segítségével könnyedén átméretezhetjük képünket. A forrás és célképek megadása után már csak az átméretezés módját kell megadni egy konstans segítségével.
Egy másik, a grafikus programokban gyakran használt művelet az elmosás, amihez a cvSmooth használható. Megadjuk a forrást, célt, valamint az elmosás típusát (pl: CV_GAUSSIAN, CV_MEDIAN). Az utolsó négy paraméterrel szabályozhatjuk magát az algoritmust.
Az utolsó két ismertetésre kerülő függvény közös jellemzője, hogy egy úgynevezett konvolúciós kernelt használ a kép módosítására. A kernel előállítására a cvCreateStructuringElementEx-t használjuk. Ez egy egyszerűsített módszer a kernel létrehozására. Ahelyett, hogy a mátrixot elemek szerint adnánk meg (bár erre is van lehetőség), csak a méretét, alakját és a kernel horgonypontjait (anchor point) állítjuk be. Amennyiben elég profik vagyunk, az utolsó paraméterrel mi magunk is beállíthatunk egy kernelt.
Az így elkészített kernelt azután felhasználhatjuk a cvErode és a cvDilate függvényekhez. Anélkül, hogy a részletekbe merülnék, ez a két függvény egyfajta zajszűrőnek tekinthető. A cvErode segítségével a kisebb hibákat (foltokat, karcolásokat) tüntethetünk el, míg a cvDilate segítségével a struktúrális különbségek jobban kiemelhetőek.
Rajzolás
Új elemeket is felvehetünk a képünkre. A legegyszerűbb alakzat, amit elhelyezhetünk a képünkön, a vonal. A cvLine segítségével nem csak a színt, de a vonal vastagságát és stílusát is beállíthatjuk. Nagyon hasonló a négyzeteket rajzoló függvény (cvRectangle) paraméterezése is.
Bináris képek
Ebben a fejezetben a kép szegmentálásával foglalkozunk. Ezek alacsony szintű műveletek lesznek, nem próbáljuk meg felismerni a képen található elemeket, csupán színek alapján elkülönítjük az egyes területeket. A feltételeknek megfelelő régiók fehér színűek lesznek, míg amelyek nem felelnek meg, feketék. Ha színes képpel dolgozunk, akkor az cvInRangeS függvényt használhatjuk. Segítségével felállíthatunk két színkorlátot és minden, ami azon belülre esik, fehérként jelenik meg a cél képen.
Érdemes megjegyezni, hogy sokkal realisztikusabb eredményt kapunk, ha nem RGB, hanem HSV színtérbe konvertáljuk képünket. Nekem az a tapasztalatom, hogy nagyon nehéz olyan szűrőt készíteni, amelyik egy színt és a hozzá közel eső árnyalatokat is nagy pontossággal kiszűri.
Szürkeárnyalatos kép esetén a cvThreshold vagy cvAdaptiveThreshold függvények használhatók. Előbbi a pixelenként hajtja végre a műveletetet, utóbbi pixel blokkonként. Mindkét függvény elég rugalmasságot biztosít, hogy miként kívánunk szűrni. A következő példaprogram ezt szemlélteti a webkameránk képével:
#include <opencv/cv.h>
#include <opencv/highgui.h>
#include <stdio.h>
int type = CV_THRESH_BINARY;
void processImage(IplImage *raw, IplImage *gray){
cvCvtColor(raw, gray, CV_RGB2GRAY);
cvThreshold(gray, gray, 128, 255, type);
}
int main(){
CvCapture *capture;
IplImage *frame;
IplImage *gray;
int run = 1;
capture = cvCaptureFromCAM(CV_CAP_ANY);
cvNamedWindow("win", CV_WINDOW_AUTOSIZE);
frame = cvQueryFrame(capture);
gray = cvCreateImage(cvGetSize(frame), IPL_DEPTH_8U, 1);
while(run){
frame = cvQueryFrame(capture);
processImage(frame, gray);
cvShowImage("win", gray);
switch(cvWaitKey(10)){
case 97:
type = CV_THRESH_BINARY;
break;
case 115:
type = CV_THRESH_BINARY_INV;
break;
case 100:
type = CV_THRESH_TRUNC;
break;
case 102:
type = CV_THRESH_TOZERO;
break;
case 103:
type = CV_THRESH_TOZERO_INV;
break;
case 27:
run = 0;
}
}
cvReleaseCapture(&capture);
cvDestroyWindow("win");
cvReleaseImage(&gray);
return EXIT_SUCCESS;
}
Az a,s,d,f,g billentyűkkel változtathatjuk a cvThreshold paraméterét. ESC-re a program kilép. A következő fejezetben az előfeldolgozott képeken fogunk tovább műveleteket végrehajtani: kontúrvonalakat keresünk, időjárás bemondóvá válunk és kiváltjuk Aha-t egy programmal.
Szólj hozzá!
Címkék: programozás opencv
Az ember, aki mindenhez ért
2012.09.12. 22:42 Travis.CG
Egyik nap elkövettem azt a hibát, hogy céges emblémával ellátott pólóban mentem dolgozni. A tömegközlekedésen megkérdezte tőlem egy úr, hogy tényleg ott dolgozok-e, amiből rögtön egy bioinformatikával összefüggő beszélgetés alakult ki.
Az úr elmondta, hogy annak idején ő is biotechnológiával foglalkozott, de csak a chip technikákkal van közelebbi ismeretségben. Arra volt kíváncsi, hogy azóta milyen új trendek vannak. Meséltem neki az újgenerációs szekvenálásokról, a bioinformatikai kihívásokról, ő meg arról, hogy szeretné frissíteni tudását a biotechnológia területén.
- Járatok egy újságot is, azt olvasom, hogy szinten tartsam magam.
- Mi a címe?
- Valami Biotech. Nem ismered? - nem ismertem.
Elmesélte, hogy dolgozott rendszergazdaként, ezért ért a számítógépekhez, de szakmáját tekintve élelmiszermérnök. Jelenleg pedig biztosítási ügynök. Bár többször hangsúlyoztam, hogy én "száraz" biológiával foglalkozom, nagy élvezettel mesélte, hogy ők biztosítanak ultracentrifugákat is. Miután a biotechnológiáról áttértünk a biztosítások területére, egy nyugtalanító érzés kezdett elhatalmasodni rajtam. Mikor már az e-mail címem után érdeklődött, megemlítettem neki, hogy ha az a terve, hogy elhalmoz biztosítási ajánlatokkal, akkor felejtse el.
Azt is említette, hogy be kellett vinnie laptopját a Budapesti Műszaki Egyetemre, mert valami hekker feltörte a Linuxát. Ott viszont nem tudtak segíteni rajta. Mindent le kellett törölnie és újratelepíteni.
Kaptam egy névjegykártyát is tőle, ahol felsorolta az összes kompetenciáját: okleveles élelmiszertechnológus mérnök, számítástechnikai szakember, tanácsadó.
Miután megnyugtatott, hogy nem fog elhalmozni biztosítási ajánlatokkal, levelezni kezdtünk. A levelei aláírása további szakmákkal bővült: Élelmiszertechnológus, Sör és szeszipari mérnök, Számítástechnikai szakértő, Specialitás kereskedelem áru forgalmazás (ez nem tudom, mit jelent), Biztosítási és befektetési szakember, Specialitás informatikai beruházások megtérülése, Projekt vezetés IT terület. Azt hiszem, nem hagytam ki semmit.
A leveleit magyar nyelven írta, én mégsem értettem miről szólnak. Kedvenc levelem tőle a következő volt: "Van olyan azonosító ami fel ismeri az RNS sorozatokat. Elektronikus úton." Ez egy kérdés, azt akarta tudni, hogy a DNS chiphez hasonlóan RNS chip van-e...
Mielőtt pálcát törnénk felette, gondolkodjunk el azon, a környezetünkben hányan vannak, akik szegényes tudásukat valami módon álcázzák. Szerintem nem kell szégyellni azt, ha valamit nem tudunk. Azt kell szégyellni, ha a hiányosságunkat nem pótoljuk, esetleg arroganciával, mellébeszéléssel leplezzük.
Merjük azt mondani, hogy nem tudjuk! A nem tudás nem jelenti azt, hogy ostobák vagyunk, csupán azt, hogy nem áll rendelkezésre az információ. Ha viszont elhitetjük, hogy rendelkezünk a szükséges ismeretekkel, azzal másokat csapunk be.
Szólj hozzá!
Címkék: életmód filozofálás
Készülődés Function2012-re
2012.09.03. 15:23 Travis.CG
Könnyű dolgom van, mert az idei Function-re egy animációt adunk be, amit többségében Grass fog elkészíteni. Jeleztem, hogy én is szeretnék valami nyúlfarknyi munkát, ezért azt a feladatot kaptam, hogy modellezzek egy boxkesztyűt és egy géppuskát.
Modellezésben nem vagyok profi, de azért össze tudok rakni ezt-azt. Ráadásul ez egy hobbi, ahol az a cél, hogy jól érezzük magunkat.
Modellezéshez 3D Studiot szoktam használni, mert ez egy olyan eszköz, amit a minimális művészi vénával rendelkezőknek találtak ki. Legalábbis nekem az az érzésem. Az én kedvenc modellezési módszerem, hogy választok egy egyszerű alakzatot, majd addig nyújtom, csavarom, amíg olyan nem lesz, amilyet szeretnék. Közben gyakran élek azzal a lehetőséggel, hogy több alakzatot boolean módosítókkal összerakok.
A boxkesztyűnél ezt is csináltam. Vettem egy gömböt, átalakítottam a "Convert to editable Mesh" menüponttal, majd kijelöltem a vertexeket és elnyújtottam a gömböt. A hüvelykujjnak megfelelő részt az Extrude segítségével kiemeltem, majd elforgattam. Végül a vertexek mozgatásával a kívánt formára hoztam a testet.
A gépfegyvert már egy teljesen más megközelítéssel készült. Először is választani kellett egy kellően ikonikus fegyvert. Ez az M60-as lett, mert Rambo nem indul csatába egy ilyen nélkül. Szereztem róla egy referencia fotót és betöltöttem háttérnek és elkezdtem közönséges vonalakkal körberajzolni. A körvonalat azután kiemeltem térbe, amitől olyan lett, mintha egy deszkából vágtam volna ki a körvonalat. Ezután átalakítottam mesh-é a boxkesztyűhöz hasonlóan és egyes elemeket (pl. a gépfegyver csövét) lecseréltem olyan testekre, amelyek jobban illettek a végleges formához. Többnyire az új test és a referencia körvonal unióját használtam.
Nemrég viszont Grass belefutott egy olyan helyzetbe, amit nem tudott egyszerűen megoldani. Nevezetesen egy kockához hasonló formát kellett Menger-szivacs jellegű formává alakítani. Tudom, ez nem egy tiszta leírás, de igyekszem a történet csattanójának lelövését elkerülni. Napokat kísérleteztem a művelettel. A bonyolultságát ugyanis az adta, hogy egy erősen fraktál jellegű test létrehozása sok memóriát igényelt, miközben az eredeti forma is magas poligonszámú volt. Végül azt csináltam, hogy létrehoztam egy Menger szivacs inverzét, vagyis olyan téglatesteket, amelyek szemből a Menger-szivacsba helyezve kitöltik annak lyukait. Ráhúztam az eredeti testre és Boolean módosítóval kivontam a kettőt. Az eredmény nagyon látványos lett. Function-ön mindenki megnézheti.
Szólj hozzá!
Címkék: demoscene
Szolgáltatóváltás
2012.08.30. 23:31 Travis.CG
Otthon az internetet Y szolgáltató biztosította hosszú ideje. Egyik nap jött egy értesítés, hogy X szolgáltató felvásárolja Y-t, de ez engem, egyszerű fogyasztót (vagy politikailag korrekt módon megfogalmazva felhasználót) nem érint. Csupán abból fogom észrevenni, hogy Y-t X váltotta fel, hogy a havi elszámolás más színű borítékban fog érkezni, és néhány TV csatorna megváltozik.
Személy szerint elégedett voltam az internetemmel. Egy 80/25-ös sávszélesség, LAN-os kapcsolat elég volt mindenre. Még arra is futotta belőle, hogy VNC-n keresztül demót sugározzak Grassnak. Négy gép is osztozott ezen, mégsem volt soha semmi probléma.
Természetesen a szolgáltatóváltással mindez veszélybe került. Eltelt néhány hónap, majd hirtelen megjelent két szerelő, hogy Ők X-től jöttek, és átszereli a netet. Először csalásra gyanakodtam, nem is akartam beengedni a lakásba, de bizonyítani tudták, hogy nem kábeltolvajok. A szolgáltató ugyanis úgy döntött, hogy a LAN-os internetet megszünteti és kapok helyette jó kis koaxon keresztül működő szupergyors internetet. Csakhogy a szupergyors internet letöltésben tényleg szuper, de a feltöltés kevésbé az. Névlegesen 120/10. El is magyaráztam neki, hogy ez számomra nem tűnik fejlesztésnek. A szerelők, kezdték elveszíteni a türelmüket. Gondolom szerettek volna gyorsan végezni, de én még a lakásba sem engedtem be őket. Miután láttam, hogy ebből csak vesztesen jöhetek ki, hagytam, hogy dolgozzanak.
Hoztak új routert is, mert ugye koaxon jön az áldás. Lefitymálva megjegyezték, hogy az új sokkal gyorsabb lesz, mert, ami nekem van, csak 54Mbit/sec-t bír csak, míg az új 180Mbit/sec-el hasít. Nem mintha ez számítana, mert elég régi gépeim vannak, egyikben sincs gigabites ethernet. Bekapcsolták a szerkezetet, ami alapból DHCP-n ment és a wi-fi is be volt kapcsolva. Beszereltek mindent, majd az aktiválás előtt az egyik szerelő felhívta az irodát és átadta nekem a telefont. A kisasszony a vonal másik végén megkérdezte, mi a bajom a szolgáltatással. Neki is elmagyaráztam, hogy a régi hálózaton a feltöltési sebesség jobb volt.
- Ezt méréssel állapította meg?
- Nem, hanem a specifikációból. A régi 25Mbit/sec-el töltött fel, ez a mostani pedig 10-el.
- Ez a garantált feltöltési sebesség?
- Nem, a maximális.
Ezután számadatokat kezdett el darálni a telefonba, amiket nem tudtam megjegyezni, majd így fejezte be: Tehát az Őn nete gyorsabb lesz.
- Ha Ön mondja, akkor elhiszem.
Ha objektíven nézzük, akkor tényleg nem volt összehasonlítási alapom. Miután mindenbe beleegyeztem, aktiválták a netet.
- Mit mondtak? - kérdezte az idősebb szerelő.
- Azt, hogy ez a net gyorsabb lesz, mint a régi.
- Nevetséges. A LAN-os az egyik legjobb, de nekünk le kell építeni az egészet. De nem is ez a leggusztustalanabb, hanem az, hogy kész tények elé állítják az embereket - mondta. Elgondolkodtam, hogy megemlítsem-e, hogy ennek ő is ugyan úgy részese, elvégre Ő hívta fel a szolgáltatót, miután beszerelt mindent.
De az internet csak nem akart megjelenni semmi. Törték a fejüket nagyon, hogy most mi a hiba, de ötletük sem volt.
- A gépnek statikus az ip címe.
A homlokuk azonnal kisimult. Megpróbálták átállítani DHCP-re, de nem ment, ezért felajánlottam, hogy megcsinálom. Mikor megjelent a scene.hu, akkor mindenki megnyugodott. Megpróbáltam belépni a router adminisztrációs felületére, de nem sikerült. Ekkor a fiatalabb szerelő azt mondta, hogy a wifi SSID-je a jelszó. Megpróbáltam azt is, de nem jártam sikerrel. Még két-három próbálkoztam, majd megkérdeztem a másik szerelőt, hogy miként tudok belépni a routerbe.
- Azt nem lehet.
- Majd, ha elmentek, megnézzük mennyire nem lehet - gondoltam.
A neten keresni kezdtem a router biztonsági jelszava után, de azok egyike sem működött. Resetelni még nem akartam, mert hátha van valami olyan beállítás, amit nem ismerek, és utána nem lesz net. De X honlapján végül megtaláltam a megoldást. Alapból nincs se felhasználói név, se jelszó, csak klikkelni kell a loginre és már bent is vagyunk. Ugyanis van egy szereld magad opció is a cég repertoárjában, ahol megkapod az eszközöket és szenvedj velük, ha neked ez kell. Ez szimpatikus. Szépen be is állítottam mindent, ami nekem kell. Végül is a sebességen nem érzek semmilyen változást.
Szólj hozzá!
Címkék: életmód rendszergazda
Bioconductor installálás felhőre
2012.08.10. 11:39 Travis.CG
A Bioconductor egy olyan R csomag gyűjtemény, ami statisztikai oldalról támogatja a bioinformatikai munkát. Validálásnál, az eredmények kiértékelésénél rengeteg olyan eszközt ad a kutatók kezébe, amely nélkül a munkájuk minősége nehezebben állapítható meg.
Amikor erőforrás igényes munkáról van szó, és egy távoli gépen akarjuk kihasználni ezt a hihetetlen potenciált, akkor meg kell bírkózni a feladattal és egy olyan gépre is telepíteni kell a rendszert, amely - mint látni fogjuk - rendszergazdai szempontból több kihívás elé állít, mint egy helyi gép.
Első lépésben le kell tölteni magát az R csomagot. Választhatunk kész, bináris formát, de ezeket úgy fordították, hogy eleve feltételezték, hogy a rendszerünk tartalmaz X-t, Tcl interpretert és még ki tudja mennyi mindent. Ha ezek adva vannak, akkor nincs semmi gond, de ezek az én Amazonos gépemen nincsenek, és szükség sincs rájuk a munkámhoz. Tehát egyetlen lehetőség, a forráskód letöltése és fordítása.
>wget http://cran.cnr.berkeley.edu/src/base/R-2/R-2.15.1.tar.gz
Alapból az Amazonos gépek nem tartalmaznak fordítót, és egyes Linuxos instance-k saját csomagkezelő repositoryval vannak ellátva, de ezek megtalálhatóak a csomagok között. (Szemben az R-el, ami viszont nem) Az R csomagnak kell Fortran fordító is a C/C++ mellett:
>sudo yum install gcc44-gfortran.x86_64
A fortran fordító gfortran44 néven szerepel, ezért - miután kicsomagoltuk az R forráskódját - létre kell hozni egy környezeti változó, hogy a scriptek megtalálják.
>export F77=gfortran44
Ezután már konfigurálhatjuk a fordítást.
>./configure --with-readline=no --with-x=no
Nincs szükségem X-re, ezért kikapcsoltam. A másik opció további forráskódok telepítésétől ment meg.
A libxml2-dev és a libcurl4-openssl-dev csomagok viszont megtalálhatóak az Amazonos gép csomagjai között, ezért telepítsük ezeket is. Az R futtatásához nem szükségesek, de a Bioconductor használatához igen.
Ha a fordítás és a csomagok telepítése sikeres volt, akkor a további csomagokat már az R parancsértelmezőjéből futtathatjuk. Indítsuk el az R-t. Mivel a telepítőnek hozzá kell férnie néhány rendszer-könyvtárhoz, ezért mindezt root jogokkal tesszük.
>sudo R
>>source("http://bioconductor.org/biocLite.R")
>>biocLite("ShortRead")
>>biocLite("edgeR")
>>library("BiocInstaller")
>>biocLite("VariantAnnotation")
>>q()
Telepítés során előfordulhatnak problémák. A leggyakoribb ok, hogy valamilyen csomag hiányzik. Sajnos erről nem kapunk egyértelmű hibaüzenetet, csak annyit, hogy pl: xml-config not found. Ez annyit jelent, hogy nincs telepítve a libxml2-dev. Elképzelhető, hogy további csomagok is kellenek, de én a saját gépemre már annyi fejlesztői könyvtárat felpakoltam (samtools, bwa, stb telepítéséhez), hogy elképzelhető, valamit kifelejtettem.
Szólj hozzá!
Címkék: rendszergazda cloud computing bioinformatika
MorphOS, OSX dual boot
2012.08.08. 12:14 Travis.CG
Megtörtént amire nem számítottam: Apple gépet használok. Minden úgy kezdődött, hogy az Amiga Klubban kiosztottak 3 leselejtezett PPC-s Mac-et. Amolyan aki kapja-marja alapon. Mivel én nem nagyon kedvelem az almás gépeket, nem tülekedtem értük. El is meséltem Grassnak, aki először nyugodtan, majd egyre feldúltabb állapotban nekem esett:
- Te önzö állat! Szerinted másnak nem kellene egy Mac?
Igaza volt, tényleg nem jutott eszembe, hogy másnak is szüksége lehet ilyen masinára. Eltelt majd egy év, és ismét felajánottak nekem egy MDD-t. Tanulva a korábbi esetből, azonnal elfogadtam.
Ami miatt Amigás körökben népszerű a géptípus, az a MorphOS nevű operációs rendszernek köszönhető. El is határoztam, hogy Grassnak feltelepítem a MacOSX 10.5-t és a MorphOS 3.1-t. Az egyetlen elérhető dokumentáció alapján el is kezdtem a műveletet. Az almás rendszert feltelepítettem, de boot partícónak 60 helyett 64MB-t adtam, mert a doksi későbbi része is ennyit említ.
Először elkövettem azt a hibát, hogy a partíciók típusát beállítottam "Mac OS extended (Journaled)"-re. Ez azt okozta, hogy később a MorphOS több tucat partíciót látott, és mindegyiket érvénytelennek tekintette. Mikor nem használtam a Journaled formát, már nem volt ilyen gondom.
Az OSX telepítése ettől a ponttól kezdve nem érdemel említést, leszámítva, hogy miután újra indult, legnagyobb meglepetésemre maga a ház kezdett el zenélni. Mivel éjjel végeztem a telepítést, rendesen meg is ijedtem.
A második lépés a MorphOS telepítése volt. Az oprendszer automatikusan felismeri a partíciókat és csatolja is azokat, miután felállt a rendszer a CD-ről. Tehát a doksi nem említi, de le kell csatolni őket, ismét felcsatolni, az ott leírt paramétereket beállítva, és csak ezek után tudjuk formázni a partíciókat. A többi lépés megegyezik az ott leírtakkal.
Az operációs rendszer választás bekapcsolás után az Alt billentyű lenyomására jön elő. Ha pedig optikai meghajtóról akarunk bootolni, akkor a c-t kell lenyomni.
Szólj hozzá!
Címkék: amiga
Az igazi tudomány
2012.07.25. 16:08 Travis.CG
Be kell vallanom, elfogult vagyok. Elfogult vagyok a tudománnyal szemben. Ha meg akarják sérteni, akkor igyekszem a védelmére kelni. Komolyan gondolom, hogy a megismerés jelenleg legjobb módja a tudományos gondolkodás. Ennek egyik pillére a megismételhetőség.
A megismételhetőség egy igen sarkallatos pontja a vizsgálatoknak, hiszen egyrészt új vizsgálatok melegágya, másrészt a régi ismeretek megerősítése.
Munkám során azt a feladatot kaptam, hogy ismételjem meg ennek a cikknek az elemzését. A cikk röviden arról szól, hogy kifejlesztett a Stanford egyetem egy kísérletes és egy számítógépes módszert, hogy meghatározzák a HLA genotípusokat. A szekvenciák egy része elérhető az SRA-n, így nekem csak a számítógépes módszerrel kellett dolgoznom.
A readeket Blast-al illesztették, majd szűrték. Az illesztés és szűrés viszont nekem teljesen más eredményt hozott. Feltételeztem, hogy a szűréssel rontottam el valamit, de szűrés nélkül sem kaptam meg azokat a találatoka, amelyek a cikkben közölt eredményekre vezetnek. Adva van a válasz, hogy a Blast futtatás a hibás. Naívan írtam az egyik szerzőnek, hogy szeretném kérni a Blast program paramétereit. Mikor nem kaptam választ, írtam a következőnek. Így írtam a leveleket, egyiket a másik után, amíg el nem fogytak a kontaktok. Mikor már mindenkinek küldtem levelet, de sehonnan nem jött válasz (pedig korábban az egyik szerző pár napja levelezett is egy munkatársammal), akkor lépett a főnököm. Neki már válaszoltak. Közölték, hogy a Blast paramétereket nem adják oda! Azok ugyanis az ő intellektuális termékeik.
Az még hagyján, hogy nekem nem válaszolnak, hiszen ki vagyok én? Nincs a nevemben CEO, BMW, KGB, stb.. De hogy nyíltam szabotálják egy vizsgálat megismétlését, az már nagyon nem járja. Ezek után miért gondoljam, hogy a módszerük jó? Azért mert ők a Stanford? Ez nekem kevés.
Elképzelem, hogy Mendeltől megkérdezik:
- Hogyan kapta ezeket az eredményeket a borsóval?
- Megmondhatnám, de akkor meg kell, hogy öljelek.
Ezek után nem lenne genetika, nem lenne Watson-Crick és nem lenne nyomorult Stanford hülye paraméterekkel.
Szólj hozzá!
Címkék: filozofálás bioinformatika
Maven bonyodalmak
2012.07.19. 09:29 Travis.CG
A programozásnál nem elég a nyelvi finomságokat ismerni. A jó programozónak eszközöket is ismerni kell, amelyek meggyorsítják a munkafolyamatait. Alapvetően az én munkámat el tudom látni egyetlen vim szövegszerkesztővel, de mikor felvettek jelenlegi munkahelyre - ahol a programozók hatalmas multinacionális cégeknél dolgoztak korábban - bizony a vim már nem volt elég. (A java egyébként sem az a nyelv, amin olyan egyszerű pusztán egy szövegszerkesztővel fejleszteni. )
Szerencsére a nagyobb tapasztalattal rendelkező kollégák beüzemelték ezeket a rendszereket, és nekem nem kellett mást csinálni, csupán használnom azokat. Az idők során újabb eszközök jelentek meg, azokat integrálták, megoldották, hogy a sok inkombatibilis eszköz valami módon együtt tudjon dolgozni.
De történt, hogy új gépet kaptam, ráadásul tesztelnem kellett egy olyan gyorsan iramban fejlesztett alkalmazást, hogy elkerülhetetlenné vált, hogy ne nekem kelljen fordítani az egész rendszert. De ehhez be kellett üzemelni például a Mavent. Gondoltam ez jó alap lesz, hogy megismerjem kicsit jobban ezt az eszközt, ezért saját magam kezdtem el beállítani a gépemen.
Ahogy lenni szokott, problémába ütköztem. Megkértem a legközelebbi programozót, segítsen, de azt válaszolta, ő nem ért a Mavenhez, ez az eszköz rossz, bezzeg régen milyen jól ment a munka, amikor még XY eszköz volt a maga primitívségében. Nem volt mit tenni, szólni kellett a Maven Guru-nak, aki az egész rendszert összerakta, hogy segítsen nekem. A Maven Guru kiíratott velem egy csomó logot, elméleteket gyártott. Mondtam neki, hogy az egyes komponensek letöltésével van probléma, mert ezt mondja a hibaüzenet. De ő továbbra is üzeneteket dekódoltatott velem, kulcsokat hasonlítottunk össze, meg minden-féle feketemágiát űztünk, talán csak csirkét nem álldoztunk. Végül kiderült, hogy van egy weboldalunk, ahol egyetlen linkre kattintással kapásból a legfrissebb verziót tudjuk fordítani.
Végül nem állítottam be semmit, hanem ezt a módszert használtam. De sajnos utólért a végzet. A fejlesztést átvariálták, de a klikkelős módszert nem. Mondanom sem kell, hogy ez is a nyári szabadságok közepén ért. Mivel senki nem fogja helyettem megcsinálni, ideje, hogy kézbe vegyem a dolgot. Félretettem minden munkát, és módszeresen végignéztem, miért nem találja a Maven a titkosító kulcsot. Öt perc alatt megtaláltam, hol írtam el a fájlnevet, ami senkinek nem tűnt fel, majd elkezdtem fordítani.
Mivel a Maven előszeretettel tölt le hihetetlen mennyiségű jar állományt az internet különböző zugaiból, míg megírtam ezt a kis bejegyzést, addigra le is fordult a program.
Szólj hozzá!
Címkék: programozás
OpenCV, avagy mit csináljon az a csapat, amelyiknek nem tagja Aha? (1. rész)
2012.07.16. 14:40 Travis.CG
Amikor először hallottam, hogyan készült a Moove című Fresh!Mindworkz produkció, nagyon elcsodálkoztam. A felvételt ugyanis Aha kézzel processzálta. Minden egyes képkockánál megjelölte, hol van táncos keze. Már pontosan nem emlékszem, mennyi klikkelést kellett végrehajtania, de nekem már az egy százaléka is sok lett volna.
Felvetődik a kérdés, mit csináljanak azok a demócsapatok, ahol nincsenek ilyen elhivatott tagok, csak lusta kóderek? A válasz egyszerű: bízzuk ezt a munkát is számítógépre.
Az OpenCV egy olyan API, ami rengeteg hasznos módszerrel igyekszik segíteni a képfeldolgozást. Nem kell nekünk megvalósítani a bonyolult algoritmusokat, hanem csak használni azokat. Miként használhatjuk ezt a demosceneben? Csak nézzünk meg néhány ASD demót! Ők már 2006 óta használják olyan produkciókban, mint az Evolution of vision vagy a Happiness is around the bend.
Mielőtt elkezdeném tárgyalni, mit is tudunk kezdeni ezzel az API-val, kicsit részletezem saját tapasztalataim alapján, hogy mit is várhatunk el a számítógépes látástól. Közhely, hogy a számítógép csak pixel információkat dolgoz fel, míg az ember formákat, de nagyon könnyű erről megfeledkezni. Ha nem akarunk kedvenc videónk feldolgozása alatt kiguvadó szemmel meredni a monitorra, miközben azt üvöltjük: Miért nem látod azt az intenzitás változást! Akkor érdemes megjegyezni a következőket:
- Ami az ember számára egyforma színűnek tűnik, az a gép számára lehet eltérő. Például készítettem egy felvételt homogén fal előtt, de az árnyékom halványan rávetült a falra, a program már másik színnek látta.
- Ha csökkentjük programunk érzékenységét, olyan helyek is egyformává válhatnak, amiket mi szemmel meg tudunk különböztetni. Például a Livin' in a box című demónk készítése közben az egyik jelenet felvételénél a távolban volt egy fa, aminek az árnyéka olyan sötét volt a program számára, mint a ruhám. Ez zajként jelentkezett.
- A színeket a számítógép több komponensben látja (pl. RGB vagy HSL), ami a feldolgozásnál meglepetéseket okozhat. Pl. egy alkalommal zöld korongokat akartam detektálni, minden mást kiszűrni. Azt gondoltam, elég a zöld csatornával játszanom, de a ruhámon egy sárga minta folyton bezavart. Egyébként sok algoritmus az OpenCV-ben csak szürke árnyalatos képpel működik.
- A kamera fényérzékenysége eltér szemünkétől. Ez szintén trivialitás, de ha arra kényszerülünk, hogy mobiltelefon felvételt dolgozzunk fel, ne lepődjünk meg semmin.
- A video kódolás hibákat okozhat a feldolgozásban. Ez sem rakéta-tudomány, de az OpenCV nem minden telepítés esetén olvas minden fájl formátumot. Esetenként konvertálnunk kell, ami megváltoztathatja a feldolgozandó pixeleket.
Most, hogy túlvagyunk az "én szóltam" részen, lássuk, mit képes olvasni az OpenCV? Alapvetően a következő képformátumokat támogatja: BMP, JPG, PNG, TIFF. A videoformátumok már rendszerfüggőek. Windows alatt mindent olvasni tudunk, amihez van Video for Windows kodek. Linux alatt a dokumentáció az ffmpeget említi, de Ubuntu és Fedora esetén olvassa a GStreamer támogatta videókat. Mac alatt be kell érni QuickTime-al.
Ha valós idejű képfeldolgozásra adjuk a fejünket, akkor használhatunk webkamerákat. Jó eséllyel nem lesz gondunk vele, ha a rendszer tudja kezelni. Sok tapasztalatom nincs ez ügyben, a laptopom beépített webkameráját kezeli mindenféle hókuszpókusz nélkül.
Az OpenCV tartalmaz még egy kicsi, lebutított keresztplatformos grafikai felületet, amit HighGUI néven szoktak emlegetni. A lényege, hogy platformfüggetlen módon egyszerű felületeket fejleszthetünk. Az eszközkészlete elég szegényes, csak ablakokat, képeket és csúszkákat hozhatunk létre.
Ennyi bevezető után lássunk egy kis kódot, ami alig csinál valamit, de ismerkedni jó lesz:
#include <opencv/cv.h>
#include <opencv/highgui.h>
#include <stdio.h>
void processImage(IplImage *raw){
cvSmooth(raw, raw, CV_GAUSSIAN, 17, 0, 0, 0);
}
int main(){
CvCapture *capture;
IplImage *frame;
IplImage *proc;
int key;
capture = cvCaptureFromCAM(CV_CAP_ANY);
cvNamedWindow("win", CV_WINDOW_AUTOSIZE);
while(1){
frame = cvQueryFrame(capture);
processImage(frame);
cvShowImage("win", frame);
if(cvWaitKey(10) != -1) break;
}
cvReleaseCapture(&capture);
cvDestroyWindow("win");
}
A kód a következő módon fordítható GNU/Linux rendszeren:
gcc simplecapture.c `pkg-config --libs opencv`
Végezetül a könnyed műfajok kedvelőinek itt van néhány tipp, miként rejtőzködjünk el a kamerák arcfelismerő algoritmusai elől divatos, de extravagáns frizurákban: CVDazzle
Szólj hozzá!
Címkék: programozás opencv
Jó, jó, de nekem Windowsom van
2012.07.14. 19:49 Travis.CG
Nemegyszer előfordult, hogy biológusok, orvosok tanácsot kértek tőle, hogyan végezzenek el egy bizonyos elemzést. Régebben elmondtam a program nevét, felsoroltam a paramétereket, amikor váratlanul megkérdezték
- Tudni fogom, hova kell klikkelni?
- Persze. Sehova. Ez egy parancssoros program.
Ekkor derül ki, hogy a kérdező valami Windowsos programot szeretne, ahol csak klikkel egyet és megkapja az eredményt. Mindezt lehetőleg egy átlagon aluli teljesítményt nyújtó számítógépen. Ilyenkor természetesen nem tudok érdemben segíteni, legfeljebb felajánlom, hogy majd lefuttatom én a programokat egy GNU/Linuxos gépen.
Később már visszakérdeztem, milyen számítógépeken akarnak bioinformatikát művelni, amivel a félreértéseket elkerülhettem. Azért felmerült bennem, mégis milyen lehetőségek vannak Windowson? Ebben a posztban kipróbálok néhányat ellenséges operációs rendszeren.
Tesztkörnyezet
A tesztgép egy AMD 4400-as 2,2GHz-en, 4GB memóriával. Az operációs rendszer 64 bites Windows 7, amire a cikk idején az összes automatikus frissítés felment. A webes megoldásokkal nem foglalkoztam, csak olyan programok próbaverzióit használtam, amit bárki le tud tölteni. (Mint az látszani fog a posztból, ez nem is mindig olyan egyszerű)
A tesztfeladat elég egyszerű: Egy Ion Torrent szekvenciát kell a humán referenciához illeszteni, megkeresni a variációkat, és összevetni egy már ismert eredménnyel. A szekvencia nyilvánosan elérhető, de sajnos regisztrálni kell az oldalra. A szekvenáláshoz egy AmpliSeq kitet használtak, ami több, daganatos megbetegedésekben szerepet játszó genetikai polimorfizmusok környékére szűkíti a szekvenálást. Az oldalon a szekvencia mellett megtaláljuk a szekvencia célpontokat (BED állomány), valamint egy másik analízis eredményét (VCF).
Geneious 5.6.4
A Geneious 14 napos próbaverziója könnyen letölthető. Még valódi e-mailt sem kell megadni. A .gz kiterjesztésű fájlokat elkezdte betölteni, de egy idő után rájött, hogy nem tudja, mit is tartalmaz, ezért nem ment tovább. A kibontás után, importálás közben automatikusan meghatározza a fájl típusát. A referencia fájlokkal könnyedén boldogult, még arra is rákérdezett, hogy a sok kijelölt FASTA állományt egybe kezelje-e.
Az illesztésnél már adódtak problémák. A teljes humán referencia futtatásához 8Gb-ra van szükség, holott minden egyes kromoszóma külön-külön beleférne 4Gb-ba. Az alapértelmezett memória, amit a program fel tud használni 1Gb, amit nekünk kell manuálisan megnövelni. (De ehhez adminisztrátori módban kell futtatni a programot.)
A BED állományt is szépen beolvasta, de előtte ki kellett jelölni, hogy melyik referenciát kívánjuk annotálni. Ez általánosságban jellemző a programra, hogy először mindig az adatokat kell kijelölni, és csak utána kell kiválasztani a funkciót.
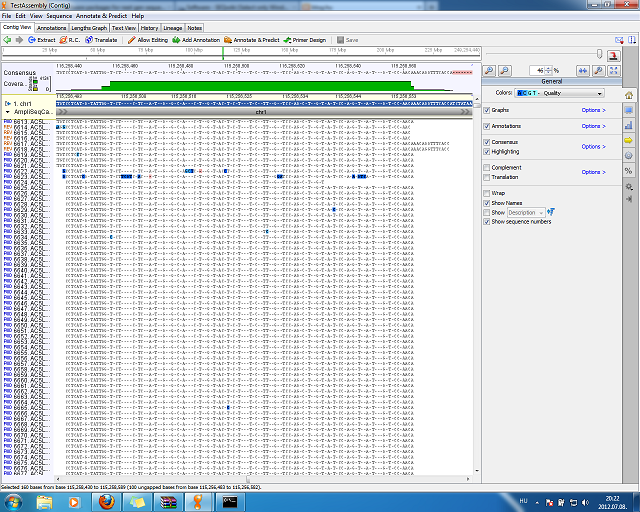
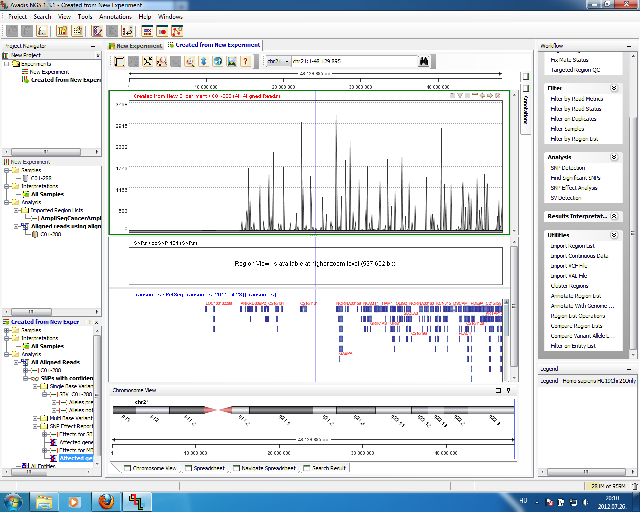
Az illesztés megtekintésekor ellenben speciális menüket kapunk, hogy a további vizsgálatokat elvégezhessük itt. A nukleotid polimorfizmusok keresését a kissé megtévesztő Annotate & Predict menüpont alatt találjuk. Az eredményeket pedig CSV állományba menthetjük. Nem VCF, de azért megteszi. Az viszont már gond, hogy exportáláskor az összes annotáció egyszerre kerül mentésre, tehát a variációk és az amplifikált pozíciók ugyan abban a fájlban lesznek. Az illesztés is exportálható SAM formátumban.
Mivel a VCF-t nem tudtam importálni, ezért nem tudtam összevetni sem, amit kaptam a teszt adatokkal. Az amplifikált régióban található variációkat is csak szemmel tudtam azonosítani, ráadásul az exportálás során összemossa az annotációkat. Ugyanakkor pozitívum, hogy a táblázat elemei között klikkelve az illesztés megfelelő pozíciójára ugrik, ami segít.
Összességében ez a program elég sok manuális munkát ad. Nehézkes, az egyes funkciók eléréséhez felugró ablakok környezetfüggő menüiben kell matatni. Továbbra is az a véleményem, hogy klasszikus feladatokhoz remekül használható, de újgenerációs szekvenálások esetén inkább kerüljük el.
CLC Genomics Workbench 5.5 beta 2
Úgy döntöttem, mivel nemsokára kijön az új verzió, ezért a bétával fogom a teszteket lefuttatni. Kérnek regisztrációt, de semmi valódi adatot nem kell megadni.
A referencia importálása kromoszómánként történt, és a program kromoszómánként is kezeli őket. Nincs olyan lehetőségünk, egy egybe fogjuk őket, mint a Geneious esetén. Természetesen egy könyvtárba szervezhetjük őket, de az mégsem ugyan az. A FASTA állományok importálásával nem volt semmi gond, leszámítva, hogy sokáig tartott.
A readeket szekvenáló gép alapján lehet importálni, az Ion Torrent támogatott, nem volt vele probléma. Az annotációval annál inkább. Sem BED, sem VCF állományokat nem tudtam betölteni, de a GFF támogatott.
Illesztésnél megadhatunk több referencia fájlt, és nem akarja egyben betölteni az összeset a memóriába. A beállítások egyszerűek egy parancssoros programhoz képest. Bár most nem használtam, lehetőség van egyszerűbb kötegelésre is, ha több szekvenciát is szeretnénk hasonló beállításokkal futtatni. Nem túl bonyolult, de adott esetben hasznos is lehet. A kész illesztést elmenthetjük SAM/BAM formátumban.
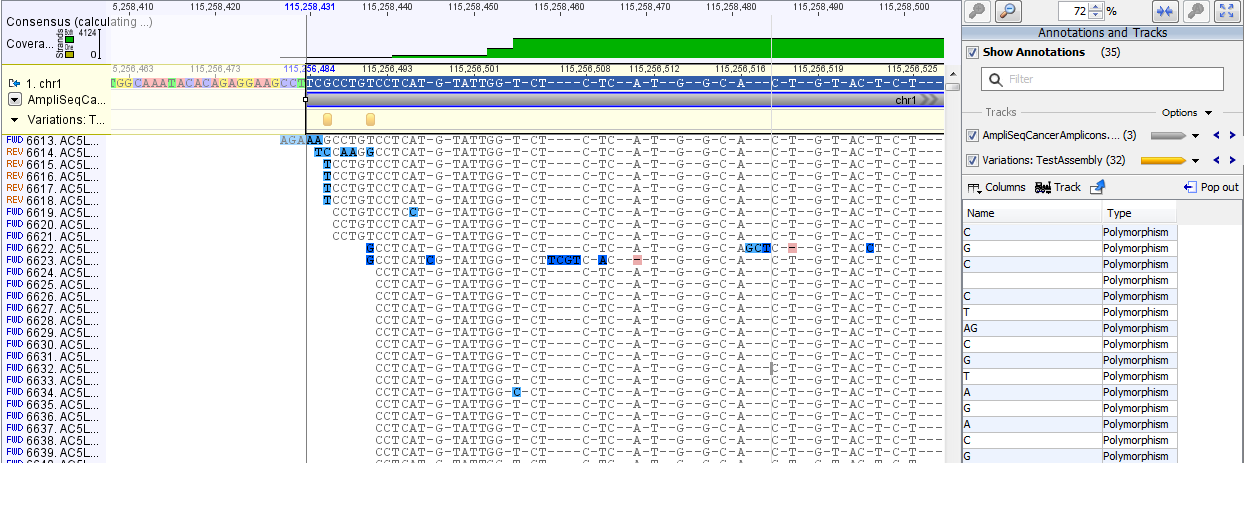
A variációk felderítésére két módszer is van. Az egyik a nemrég debütált Probabilistic Variant Detection, míg a másik, a hagyományos alapokra (a referenciától eltérő readek arányán alapul) épülő Quality-based Variant Detection. Az Ion Torrentre jellemző homopolimer hibát képesek az algoritmusok figyelembe venni. Az eredmény mindkét esetben lehet annotációként a referenciára rakni, vagy külön táblázatban nézegetni. A Geneiousnál megszokott klikkeléssel nem lehet a referencia megfelelő pontjára ugrani, azt kézzel kell kimásolni (a vágólapot sem használhatjuk a koordináták másolására). A táblázatot CSV formátumban is elmenthetjük.
Az eredményt annotációs trackként is megjeleníthetjük. Ez, bár nem egy szofisztikált eszköz, de használhatjuk a variációk összehasonlítására. Mivel VCF-t nem tudtam betölteni, ezért a kétféle variáció detektáló algoritmus eredményét hasonlítottam össze a képen. Az elemzésben több segítséget nem kapunk.
A CLC népszerű újgenerációs szekvenálások feldolgozásában. Rengeteg eszközt ad a kutatók kezébe, ha kis számú adattal kell szöszmötölni. A számítógép mellé persze nem árt egy hagyományos jegyzetfüzet, mert a vágólap használata nélkül kénytelenek vagyunk kézzel lejegyezni apró információkat.
GeneSpring NGS 12.1
A GeneSpring, mikor még aktívan használtam, egy chip elemző program volt csupán. Azóta ők is nyitottak az NGS felé. A próbaverziót regisztráció után lehet letölteni, ahol bármilyen e-mail címet megadhatunk. Mivel már korábbról volt hozzáférésem, ezért könnyedén le is tudtam tölteni a próbaverziót, amit 20 napig használhatunk.
Sajnos a licencet már nem sikerült szereznem hozzá, mert 6 évvel ezelőtt letöltöttem egy másik próbaverziót, ezzel eljátszottam minden életemet. Később sikerült aktualizálni a licencet, így már nyugodtan kipróbálhattam a programot.
Az első kellemes meglepetés az annotációk automatikus frissítése volt. Az első indítás után kiválaszthattuk az általunk vizsgált lény referenciáit és beállíthatjuk, hogy mit töltsön le. A szokásos referencia importálást ezért kihagytam és a frissítést választottam. Kiválasztottam a dbSNP-t is, ami jelentősen megnövelte a letöltési időt. Nagyjából 4 órán át tartott a RefSeq, a genom és a 135-ös dbSNP importálása, amit én soknak tartok.
Fontos kihangsúlyozni, hogy a Genesprint az Agilent terméke, ami azért fontos, mert a szokásos DNS chipek mellett más laboratóriumi kiteket is forgalmaznak. Ezen kitek kiértékeléséhez szükséges egyéb adatok a programba be vannak építve. Például a SureSelect targetáló termékcsalád genomi célpontjai.
A program nem tartalmaz short read import funkciót. Egyáltalán semmit nem tudunk kezdeni short readjeinkkel ezzel a programmal. Az illesztés ki-ki oldja meg maga saját eszközével. A BAM fájlt már be lehet tölteni és lehet keresni nukleotid polimorfizmusokat.
Sajnos nem találtam meg a módját, hogyan lehetne VCF fájlokat beimportálni, pedig ha sikerült volna, akkor a Differential SNP Analysis segítségével izgalmas összehasonlításokat lehetett volna csinálni. De ha BED vagy CSV formátumban vannak adataink, akkor mindez nem jelent problémát.
Egyetlen komoly problémám volt csak a programmal, hogy rettenetesen lassú volt. Minden egyes művelet esetén valamit nagyon töltött. Még ha csak a látható ablakokat akartam kimenteni a program beépített képlopó módszerével, akkor is elkezdett tölteni, frissíteni. Ha kicsit átméreteztem az ablakokat, akkor is elkezdett újrarajzolni mindent, amihez töltött valamit a merevlemezről. Az a gyanúm, hogy semmilyen indexelést nem végez a program, ezért minden újrarajzolás idején elkezdi betölteni a referenciát és az annotációt, hogy megkeresse a kirajzolandó adatokat.
A program sokat tud, de nehéz eligazodni rajta. A súgó pedig leírja, hogy miként kell egy analízist elvégezni, de a hibakezelés teljes egészében hiányzik. Néhányszor előfordult, hogy a súgó szerint követtem a lépéseket, mégis kaptam egy hibaüzenetet, amivel nem tudtam mit kezdeni.
Az előző két programhoz képest itt nem műveleteket végezhetünk, hanem elemzéseket. Nincs illesztés, nincs primer tervezés, van viszont R integráció, SNP feldolgozás és különböző összehasonlítások. Statisztikát kapunk mindenről, amiről csak akarunk.
Avadis NGS 1.3.1
Már a poszt megírása után tudtam meg, hogy létezik még egy program, ami érdemes lehet, hogy belevegyem az összehasonlításba. Ez a program nem más, mint az Avadis. A felhasználói felület és a program logikája teljesen olyan, mint a GeneSpringnek. Nem tudom, mi a kapcsolat a két cég között, de az biztos, hogy azonos kódbázisból dolgoznak.
A két program azonban teljesen más felhasználási területre készült. Amit hiányoltam a GeneSpringből, azt itt megkapom. Először is, lehet közönséges FASTQ fájlokat betölteni, akár tömörített formában is. A readek minőségéről kapunk statisztikát, mintha a FastQC-t használnánk. A BED állományt is minden gond nélkül be tudtam tölteni.
A readek illesztéséhez a BWA-ból ismert Burrows-Wheeler alapú illesztőt használ. Arról, hogy mennyire jó, talán majd egy másik bejegyzésben írok. Az illesztés minőségéről szintén képet kaphatunk néhány statisztika segítségével. Ez szintén hasznos funkció, és nem is láttuk másik alkalmazásban.
Szemben a GeneSpringgel, itt már tudunk VCF állományokat is importálni, valamint többféle variáció felderítő algoritmussal is kísérletezhetünk. A hagyományos SNP keresésen kívül felderíthetjük a strukturális változásokat, az SNP Effect Analysis segítségével pedig meghatározhatjuk, az adott variációnak milyen hatásai lehetnek. A különböző variációkat össze is hasonlíthatjuk, ezzel ez volt tesztünk egyetlen programja, ami mindegyik feladatot maradéktalanul végrehajtotta.
Lasergene Genomics Suite
A DNAStar programcsomagot régen használtam, méghozzá azt a verziót, amit minden magyar kutatóintézet használt. Egy PhD-s, aki fiatal korában egy külföldi cracker csapat tagja volt, feltörte a programot, és attól a pillanattól kezdve mindenhol azt használták. Már megjelent több újabb változata is a programnak, de itthon mindenki csak ezt a régi, de "ingyenes" verziót használta, mivel a benne található funkciók bőven kielégítették egy átlagos kutató igényeit.
Az új divathullámnak megfelelően van NGS alapokon nyugvó programcsomagjuk is, amit egyszerű halandó nem tölthet le, csak, ha regisztrál. A regisztrációt én nagyon komolyan szoktam venni, ezért olyan nevet szoktam választani, amiről lerí, hogy semmi köze a valóságoz. Az űrlap a végén arról tudósít, hogy egy munkanapon belül megkapom a letöltéshez szükséges információkat.
Sajnos ez nem igaz. Valóban kapok e-mailt. de arról, hogy válaszoljak olyan kérdésekre, mint például mennyi pénze van a cégnek, mi a kutatási területünk, stb. Itt kicsit felhúztam magam, ezért megírtam, hogy nem vagyok hajlandó egyetlen kérdésre sem válaszolni, amíg meg nem kapom a próbaverziót, valamint nem értem, hogy ez a bonyolult rendszer miért szolgálja az én érdekeimet. Válaszoltak, ahol leírták, hogy meg akarják védeni a programjukat az olyanoktól, mint én, akik 30 napig ingyen használják azt a napi munkájukhoz, de utána nem fizetnek. Megírtam, hogy ehhez a blogbejegyzéshez kellett volna a program, de mivel ez egy kicsi, magyar nyelvű blog, senkinek nem fog hiányozni a DNAStar terméke.
Partek
Ez az a program, amiről a poszt írásának idején értesültem először. Egy Google keresés adta ki eredményül. A próbaverzió megszerzése kisebb hadműveletet igényel. Először is egy webes űrlapot kell kitölteni, ahol a Hotmailt. GMailt és társait nem adhatjuk meg e-mail címként, csak céges e-mailt. Mivel még működik a régi egyetemi címem, azt adtam oda nekik. Kaptam egy levelet a következő munkanapon, ahol egy lmtools.exe letöltésére és futtatására szólítottak fel. Ez a gépről egy csomó adatot összeszed, becsomagolja, majd vissza kell küldeni a válasz e-mailben. Csak ezután kapjuk meg a licencet. Én egy hét után sem kaptam tőlük semmit, pedig megpróbáltam egy másik regisztrációt is új néven.
Összefoglalás
Még mindig nehéz dolga van annak, aki Windows alatt szeretne bioinformatikát művelni. Az Avadis NGS kivételével mindig akadt egy-két lépés, amikor el kellett volna hagyni a programot, hogy a feladatot máshol oldjuk meg. De sajnos ezek a programok nem arra vannak kihegyezve, hogy kooperáljanak más alkalmazásokkal, hanem ellenkezőleg, igyekeznek elszigetelni a felhasználót a külső világtól. Kevés az import/export funkció (bár meg kell jegyezni, hogy mind a Geneious, mind a CLC bővíthető pluginekkel). Érdekes kirándulás volt ez a klikkelős világban, de ha el akarom végezni a munkámat, akkor jobban megbízom az összehekkelt Bash scriptjeimben és a parancssoros programokban. De ha valakinek mégis egy összetett alkalmazás kell, akkor az Avadis megfelelő választás lehet.
3 komment
Címkék: bioinformatika
Így készült: Livin' in a box
2012.07.04. 11:19 Travis.CG
Ez a demónk egy kísérletből született. Egy ideje nagyon foglalkoztat a házi motion capture gondolata, ezért elkezdtem tanulni az OpenCV-t (amiről hamarosan írok egy poszt-sorozatot). Úgy láttam, ez nagyon jó demo-alap lenne, Grass-al ezért elkezdtük tervezni a demót.
A másik lökést az adta, hogy tavaly megemlítettem egy AmiCon-on is, mivel foglalkozom, és Spenot felajánlotta, hogy kölcsönadja a Kinectjét, hogy azzal is kísérletezzem. Decemberben került sor az átadásra, egy hétig játszhattam vele. Gyorsan lementettem néhány videót, amit majd remélhetőleg fel fogok tudni használni a demóban.
Az egyetlen probléma az volt, hogy megközelítőleg 2GB méretűek voltak a mélységi információk. Ez még a legengedékenyebb compó-szabályzatnak sem felelnek meg. Szükség volt valami tömörítésre. Egy viszonylag egyszerű megközelítést alkalmaztam. Ahol a következő képkockában eltérés volt az előzőhöz képest, ott letároltam a koordinátát és a megváltozott értéket. Ezzel 7 és 11 MB méretűek lettek a fájlok. Úgy gondoltam további trükközésre nincs szükség.
Januárban videókat készítettem egy mobiltelefonnal, amit egy Amigás winchester beépítő kerettel rászereltem egy fotóállványra. A telefon videorögzítő képességével nem voltam megelégedve, mert gyakran minden figyelmeztetés nélkül leállt a felvétel. A másik probléma a színhűséggel volt. A fakó színek miatt a videofájlok feldolgozása volt problémás. Közben folyamatosan fejlesztettem egy kontúr detektáló programot, ami OpenCV-n alapult.
Februárban Grass nekem adta az egyik fényképezőgépét, ami már könnyedén ráült a fotóállványra, a videofelvevő képességével is elégedett lehettem. Egyedül az alacsony akkuidő hátráltatott. A kontúrokat már jól felismerte a program, el is készültek a demó felvételei. Annyit azért meg kell jegyeznem, hogy igyekeztem megkönnyíteni a program dolgát, amennyire csak lehetett, ezért fekete szabadidő ruhában, kesztyűben ugráltam a kamera előtt. Ez télen még nem volt akkora probléma (bár a hó a hátamon megolvadt), tavasszal viszont izzasztó volt.
Úgy emlékszem, ez idő tájt született meg a demo címe is, amit Grass talált ki, egy régi együttes után, akiknek hasonló nevük volt, legismertebb daluk pedig megegyezett az együttes nevével.
Márciusban a felvételekkel párhuzamosan írtam a demót, Grass pedig ellátott különböző tartalmakkal és ötletekkel a színekre vonatkozóan. Mivel elég messze lakunk egymástól, és nem volt demo kompatibilis laptopom, a gépemet megosztottam VNC-vel, így igaz, hang nélkül, de Grass is láthatta, hogyan alakul a produkció. Néhány esetben még az is ment, hogy az ötleteit azon nyomban megvalósítottam.
Néhány jelenetet leszámítva elkészült Revisionig, ahol mindenki tudja, mi történt. Parti után egy ideig nem csináltam semmit, majd ismét nekiláttam foltozni a hiányzó részeket.
Egyetlen nagyon komoly hiányossága volt/van a demónak, mégpedig a trianguláció pontatlansága. A demó ugyanis csak a körvonalat tölti be, majd felosztja háromszögekre és textúrázza a poligont. Sajnos egyes esetekben ez a lépés megszakad és üres lyukak maradnak. Rengeteg időt töltöttem, hogy megoldást találjak. Megpróbáltam egy automatikus módszerrel kiszedni a kereszteződött szakaszokat, de ezzel gyakran kivágtam a kontúr nagy részét, így inkább letettem róla. Próbáltam másik algoritmust készíteni, de azzal egy csomó időt elbíbelődtem, és nem készült el. A határidő viszont kérlelhetetlenül közeledett. Végül megpróbáltam csalni. A lyukakat valami gyors módszerrel befoltozom, de ezek általában konvexé alakították a poligonokat, ami ugyancsak rosszul nézett ki. A végén semmit sem változtattam meg.
A demóban igyekeztem OpenGL 4.1-t használni, mert így sok effektet shaderben tudtam programozni. Sokat segített, hogy nem kell újrafordítani a demót, csak a shader forrásában turkálva állíthatom a paramétereket. Ez főleg akkor volt a segítségemre, amikor a tömegközlekedésen kellett tuningolni a demón. Más racionális oka nem volt a választásnak. Egyébként is úgy szeretek demót írni, hogy valami újat tanuljak az elkészítése közben.
A produkciót mindenki megtekintheti a poue.net-en, YouTube-n. A forráskód elérhető GitHub-on. Ez utóbbinak az az előnye is megvan, hogy Gargaj elkészítette a Windowsos válozatát is. Ha lesz felvétel az élő közvetítésről róla, azt is belinkelem.
Szólj hozzá!
Címkék: demoscene
Amiga a Newtech Meetupon
2012.06.18. 09:28 Travis.CG
Charlie röviden összefoglalta a Newtech Meetupon az újgenerációs Amigák helyzetét. Az előadás kicsit spontánra sikeredett, de ez nem okozott gondot senkinek. Az előadás után megnézhettek az érdeklődők egy Efikát és egy MacBook-ot, amin MorphOS fut. Megjegyzem, ez utóbbi Magyarország egyetlen laptopja, amin újgenerációs Amiga fut.
Természetesen voltak más érdekes előadások is. Kiemelném az újra felhasználható elemekből álló műanyag préselő szerszámokat, illetve egy beágyazott rendszerek fejlesztésébe betekintést engedő előadást.
Volt egy közösségi oldalakat összefogó közösségi oldal is, aminek a létjogosultságát nem sikerült megértenem. (Bár ebben az is közrejátszhatott, hogy elég ellenségesen viseltetek a kismillió közösségi oldal irán, ami inkább elszigeteli az információáramlást, mint ténylegesen segítené, de erről egy másik posztban)
Jók ezek az összejövetelek, mert adnak egy kis kitekintést, hogy mások épp mivel próbálkoznak, milyen problémákat oldanak meg. Nekik magunkak kell kitalálni a lépéseket, mert - ahogy a mottóm is mondja - senki nem oldja meg helyettük.
Szólj hozzá!
Címkék: amiga
@Party beégés
2012.06.17. 21:50 Travis.CG
A tengeren túli partikkal az a baj, még ha közvetítik is élőben az adást, azt is egy lehetetlen időpontban lehet csak megnézni. Így volt ez az @Partyval is, amit kénytelen voltam hajnali 3-kor megtekinteni. Természetesen a compók késve kezdődtek, így a napfelkeltét a számítógép előtt néztem végig. Közven azon gondolkodtam, miért nem keltem fel később, hiszen a zenei és grafikai alkotásokra nem vagyok kíváncsi.
Ennek ellenére a vetítő compó igen érdekes volt. Régi írásvetítőt kellett használni, hogy demószerű effekteket érjenek el. Meglepő, hogy mire képesek a találékony elmék...
Engem persze a mi demónk érdekelt leginkább. Reggel 6 körül már le is adták. A szervezők a nagy partykhoz hasonlóan felvételről adták le a demónkat, ami nem lett volna baj, ha nem csúszik el a kép és a hang szinkronizációja. A másik probléma az volt, hogy a tesszelációs shaderrel készült hátterek irtó bénán néztek ki. Olyan volt, mintha a shader futása megszakadt volna félúton. A vége előtt pedig egyszerűen megszakadt.
Összességében egy katasztrófa volt. Az eredményeket még nem tudom, de nem számítok sok jóra.
Frissítés:
A pouet.net kiírás szerint úgy tűnik, mintha megnyertük volna. Ha ez így van, el sem merem képzelni, milyen lehetett a többi induló.
Szólj hozzá!
Címkék: demoscene
Minek nevezzelek?
2012.05.30. 22:00 Travis.CG
A Magyar Bioinformatikai Társaság éves gyűlésén érdekes beszélgetésnek voltam tanúja. Az utánpótlás nevelésről folytatott eszmecsere során a jelenlévők elmesélték, hogy az országban hol, milyen formában oktatnak bioinformatikát.
A Pázmány Péter Katolikus Egyetemen például molekuláris bionika néven találkozhatunk vele. Habár az oldalon olvasható leírás nem kapcsolódik szorosan ahhoz az ismeretanyaghoz, amit itt a blogomban bemutatok, nem árt tudni, hogy az oktatók között ott van Falus András és Pongor Sándor, akik a magyar bioinformatika meghatározó alakjai.
A Budapest Műszaki és Gazdaságtudományi Egyetemen is van ilyen tantárgy. Itt matematikai oldalról ismerhetik meg a hallgatók ezt a tudományt. A tematikát olvasva erősen statisztikai, adatbányászati központú oktatás folyik itt.
Szegeden is tanítanak bioinformatikát, ami a legközelebb van ahhoz, amit én is művelek. Ezen kívül még máshol is tanítanak ilyen-olyan néven bioinformatikát, de ennek a posztnak a célja nem az, hogy ezeket mind felsoroljam.
Tehát látunk három megközelítést, amelyek igen kis mértékben fednek át. A beszélgetés is azt a konklúziót vonta le, hogy magának a bioinformatikának nincs egységes arculata. Ha azt mondom, ornitológus, akkor mindenkinek egy olyan ember fog beugrani, aki füttyről ismeri meg a madarat. Ha botanikusról beszélek, akkor olyan valakit képzelünk el, aki hason csúszik a kvadrátban és gyökérgümő szőr alapján azonosítja a gazokat.
De ki a bioinformatikus? Egy matematikus, aki a szekvenciát L hosszúságú A halmaznak látja, ami {A,T,G,C} elemeket tartalmaz? Vagy egy olyan kutató, aki egész nap egy vegyszerszagú laborban dolgozik, és végül futtat egy Blastot az eredményekből?
Én nem tudom a választ, de a beszélgetés rávilágított arra, hogy mások sem. Szerencsére az akadémikusok mindenre tudnak megoldást, de ha megoldást sem tudnak, akkor szerveznek egy konferenciát. Ezért októberben lesz egy konferencia, ahol a spektrum összes tagja képviseltetni fogja magát és mindenki egy-egy előadással bemutatja, hogy miért is tartja magát bioinformatikusnak.
2 komment
Címkék: bioinformatika
Bioinformatika Magyarországon - előadások
2012.05.15. 22:13 Travis.CG
Ma tartották a Bioinformatika Magyarországon című előadás sorozatot, ami rövid betekintést nyújtott a hazai kutatóműhelyek elért eredményeibe. A megjelenő kis számú érdeklődő a Tudományos Akadémián a fehérjék vizsgálatától az elméleti matematikáig minden témakörben hallhatott érdekességeket.
Röviden, a teljesség igénye nélkül a következő részleteket emelném ki:
Patthy László azzal az érdekes megfigyeléssel kezdte, hogy csökken a bioinformatikába vetett hit. Míg korábban azt gondolták, a bioinformatika lesz a megoldás mindenre, addig olyan problémákat hagyott ez a tudományterület megoldatlanul, mint a génannotáció. A génannotáció pedig minden elemzés alapja. A MisPred egy fehérje térszerkezeti tudásbázison alapuló megoldást nyújt erre a problémára.
Simon István előadásában az volt számomra a legérdekesebb, hogy Ő már akkor bioinformatikával foglalkozott, amikor - kis túlzással élve - ez a tudományág még nem is létezett. A programozás mellett azt is tudja, hogyan kell megfogni a pipettát, mert a kísérletes munkából is kivette a részét. Legjobban az érzékeltette, mennyire megelőzte korát, hogy az általuk megalkotott térszerkezet prediktáló eljárás hátránya nem a pontosság volt, hanem az, hogy a kísérletes módszerek gyorsabban adtak eredményt!
Barta Endre két mezőgazdaságilag fontos haszonállat, a szarvasmarha és a mangalica genomjának bioinformatikai elemzéséről beszélt. Az ember számára fontos géntulajdonságok meghatározásán túl a fajtaspecifikus variációk felderítése és a szabályozó régiókban fellelhető nukleotid polimorfizmusok azonosítása a céljuk. Egyetlen dolog csalt csak mosolyt az arcomra: a szarvasmarhában azonosított géneket egér modellben tesztelik. A döntés labortechnikai szempontból logikus, de csak egy pillanatra képzeljünk el egy "jól tejelő egeret".
Harrach Balázs előadása a PhD hallgatók áldozatos munkájáról szólt. A csoport ugyanis vírusokat, vírus törzseket vizsgál, de gazdaállatokkal együtt. Minden egyes gazdaállat vizsgálatához szükség volt olyan PhD hallgatókra, akik megfelelő "hobby"-val rendelkeztek a minták begyűjtéséhez. Például így segített a barlangász a denevér minták begyűjtésével, vagy az ornitológus az avevirusok osztályozásánál. A lelkesedés olyan szintű a csapatban, hogy még a két hetes döglött tengerimalacot is felkutatták, csak, hogy mintát izoláljanak belőle.
Simon Péter előadásából nem sokat értettem. Egy elméleti matematikai megközelítést használtak járványterjedés szimulálására. A terjedést gráfokon keresztül írták le.
Miklós István szintén matematikai oldalról mutatta be a bioinformatikát, de igyekezett azt a hallgatóságnak is emészthetővé tenni. A biológiában előforduló folyamatok között vannak NP nehéz feladatok, ahol a számítási igény elképzelhetetlenül nagy. Az ilyen folyamatokat leíró modellek esetén azt is bizonyítani kell, hogy a feladat megoldható-e.
1 komment
Címkék: bioinformatika
Emboss: Grafikus programok
2012.05.10. 14:52 Travis.CG
Elérkeztünk kedvenc programcsomagunkat bemutató sorozatunk záró részéhez. Azokból a programokból válogatok, amelyek valamilyen kapcsolatban vannak a grafikus megjelenítéssel.
Az Emboss egyik nagyszerű tulajdonsága, hogy a grafikus tartalmat megjelenítheti képernyőn, de elmentheti PostScript, PNG állományokba is, ami egy cikk esetén lehet hasznos. Csak mellékesen jegyzem meg, hogy a legtöbb klikkelős, grafikus program mellőzi teljesen ezt a lehetőséget, és a kutató kénytelen képernyőképeket készíteni és valamilyen grafikus programmal levágni a szükségtelen részeket. De lássuk inkább a programokat.
abiview: A Sanger szekvenálás eredménye sok esetben nem egy kész szekvencia, hanem egy intenzitás görbe, ami azt mondja meg nekünk, hogy az érzékelő milyen nukleotidot detektál az adott pozícióban. A program segítségével megjeleníthetjük ezt. A másik hasznos funkciója, hogy képes kiolvasni a szekvencia sorrendet és elmenteni fájlba.

prettyplot: Többszörös illesztést jeleníthetünk meg vele. Mint minden Emboss program, ez is hihetetlen mennyiségű fájlformátumot ismer. Sajnos az interaktivitás terén nem jeleskedik, ezért ne számítsunk rá, hogy oda-vissza ugrálunk a különböző pozíciók között.
plotcon: A prettyplothoz hasonlóan itt is a többszörös illesztéseket vehetjók górcső alá. A különbség, hogy a program megpróbálja a konzerválódott szakaszokat kiemelni. A konzervált szakaszok méretét a wordsize paraméterrel állíthatjuk be.

plotorf: A lehetséges nyitott leolvasási kereteket képes megjeleníteni. Érdemes még megemlíteni a showorf programot, mely a plotorf szöveg alapú testvére. A -start és -stop opciókkal a leolvasási keretet határoló kodonokat adhatjuk meg. Amennyiben nem akarjuk mind a hat leolvasási keretet megjeleníteni, parancssori kapcsolókkal korlátozhatjuk, mit jelenítsen meg a program.

dotmatcher: Két szekvencia illesztésének vizsgálatakor lehet hasznos, ha a hasonló szekvencia-részletek vizuálisan is megfigyelhetőek. A program az összehasonlítandó szekvenciákat a függőleges és vízszintes tengelyen reprezentálja, míg a hasonló szakaszokhoz egy pontot rajzol. Ha a két szekvencia megegyezik, egyetlen vonalat látunk, ami a bal alsó sarokból a jobb felsőbe tart.

Szólj hozzá!
Címkék: bioinformatika
Az a bizonyos GTK-3.0 bug
2012.05.02. 14:50 Travis.CG
Tisztelt Bíróság!
Az a bug úgy volt, hogy nem is volt bug. Tetszik tudni, én írtam egy demót Revisionre, amit nem is adtak le. Azt mondták, hogy lehet 1024x768 módot támogatni, ha akarok, és én akartam. Ugyanis tisztelt bíróság, nekem nincs ilyen széles izém, mi az? képernyőm. És én csak 4:3 oldalaránnyal tudok dolgozni, és ezért lett ez a felbontás.
Aztán szóltak a compó előtt, hogy nem megy ki teljes képernyőre a demó, pedig olyan felbontása volt, amire aztatat mondták, hogy lehet. De amikor láttam a compógépen a felbontást, annak az oldalaránya nem volt 4:3, hanem inkább 16:9. És az Ubuntu 12.04 ezen a felbontáson kiteszi az ablakot teljes képernyőre, de a Unity eszköztár ott marad. De azután otthon csak annyit csináltam, hogy felvettem egy 16:9-es felbontást, és a Unity eszköztár eltűnt, amikor kiment teljes képernyőre a demó. Tehát kérem szépen, a kódban nem volt bug, csak nem volt felvéve a megfelelő felbontás. És akár mit dönt a bíróság, ez a demó le fog menni @Partyn, mert ott jó fejek az emberek.
Szólj hozzá!
Címkék: programozás
Mese a Linux hekkerekről és a vicces laptopról
2012.04.17. 22:20 Travis.CG
A Kerekerdő közepén a törpék Linuxot használnak a számítógépeiken. Sokáig használják azokat, így gyakran lemaradnak a legújabb trendekről és újdonságokról. Jól elvannak a kis parancssorukkal, lassan forgó merevlemezükkel és a szaporodó pixelhibákkal együtt.
Történt azután, hogy hogy-nem, egy program tesztelése közepette rájöttek, hogy bizony 1-2 GB gyakran nem elég az összes adat feldolgozására. Tanakodtak is sokat a kerek erdő közepén, a munka meg csak állt, mert bizony a régi laptopok nem átallottak Out of memory kivételeket dobni.
A nagy varázsló ekkor úgy döntött, hogy a bevételekből ideje áldozni szorgos kis törpéire, hogy még vidámabban húzzák az igát. Kaptak is szép új laptopokat. Csodájára járt Kerekerdő apraja és nagyja. Csak nézték a villogó ledeket, símogatták a tapipadot, ami szinte teljesen beleolvadt a burkolatba. Keresték a pöcökegeret, de ezeken az új masinákon már nem volt ilyesmi.
Nosza, a törpék rögtön elkezdték telepíteni az Ubuntut, annak is 11.10-es változatát. Közben tovább nyomkodták az érdekes és rejtélyes gombokat. A telepítés végeztével viszont furcsa jelenség zavarta meg kis törpéink életét. A tapipad nem ment! Az egérmutató ott virított a 16:9-es képernyő közepén és nem mozdult.
Ejj lett ám szitkozódás, hogy csak úgy zengett Kerekerdő! Bizony, nem voltak restek összehozni az Ubuntu fejleszőinek édes szülőit minden féle egzotikus állattal, majd ékes szóval le is festették a légyottokat.
A legbölcsbb törpe, aki máskor a káromkodásban is élen jár, most csak csendben mosolygott. Várta mikor száll el a harag köde a fiatalabb törpék szeméről, majd megsúgta nekik a megoldást. Rendszergazdakén el kell távolítani a psmouse kernel modult, majd újra be kell tölteni. Néztek nagyot a törpék, majd szó nélkül másztak be a parancsértelmezőbe és kapcsolgatták a psmouse kernelmodult, mintha mi sem lenne természetesebb. Láss csodát, a tapipad megjavult. Ismét működtek a laptopok.
Csak később vették észre, hogy ezeken a fránya új laptopokon ki lehet kapcsolni a tapipadot egy jelölés nélküli gombbal. A nagy játékban bizonyára lenyomták anélkül, hogy tudatosodott volna bennük, mit is csinálnak. De végül sikerült egy igazán bonyolult megoldással rendbehozni.
Szólj hozzá!
Címkék: irodalom
Heng Li az idei Benjamin Franklin díjas
2012.04.17. 14:20 Travis.CG
Az idei Benjamin Franklin díjas Heng Li. Röviden tekintsük át, miért is érdemelte meg ezt a díjat.
Az újgenerációs szekvenáló gépek kimenetét a legtöbb esetben egy referencia genomhoz kell illeszteni. Az illesztés kimenete egy SAM vagy BAM fájl. Amennyiben ezeket a fájlokat fel akarjuk dolgozni, az első program, ami kezünk ügyébe kerül, a Samtools. A programcsomag fejlesztése a nevéhez kapcsolódik. A Samtools jelentőségét mi sem mutatja jobban, hogy a Torrent Suite 1.5 részét képező SNP detektáló algoritmus ezen alapul.
A másik híres alkalmazása a BWA, egy rendkívül jól megírt illesztőprogram. A BWA-val sokat dolgoztam, összehasonlító tesztekben ezt tekintettük alapnak, amelyhez viszonyítva igyekeztünk jobb eredményeket elérni. (Talán annyit elárulhatok, hogy elég kevés sikerrel.) A memória igénye alacsony, sebessége pedig bőven kárpótol mindenért. Összehasonlító tesztekben még nem láttam olyat, ahol lecsúszott volna a dobogóról.
Chip-Seq kísérletek kiértékelése esetén találkozhatunk a MAQ programmal. Személy eddig még nem használtam, ezért nem is szeretnék róla sok szót ejteni.
Aki ezek után azt gondolja, hogy Heng Li csupán egy ügyes programozó, téved. A rizs genom véglegesítésében is dolgozott, csak úgy, mint a selyemlepke szekvenálásában. Ha valakinek még ez sem lenne elég, a csirke variációs térképének feltérképezésében is közreműködött.
Publikációs listája itt tekinthető meg. Innen is gratulálunk neki a díjhoz.