Az ember vizuális típus. Akkor is szereti grafikusan megtekinteni munkája eredményét, ha annak nincs is gyakorlati haszna. Így van ez a bioinformatikában is. Az újgenerációs szekvenálási eredmények grafikus megtekintése időrabló feladat. Az adatok mennyiségét figyelembe véve gyakran szükségtelen. Viszont előfordulhat olyan eset, amikor nem kerülhetjük meg azt, hogy szemünkre és józan eszünkre támaszkodva megnézzük, mi is kaptunk eredményül.
Jelen posztomban több programot fogok bemutatni, amelyek alkalmasak arra, hogy illesztéseket jelenítsenek meg. Mind kereskedelmi, mind ingyenes programok belekerültek a tesztbe.
EagleView
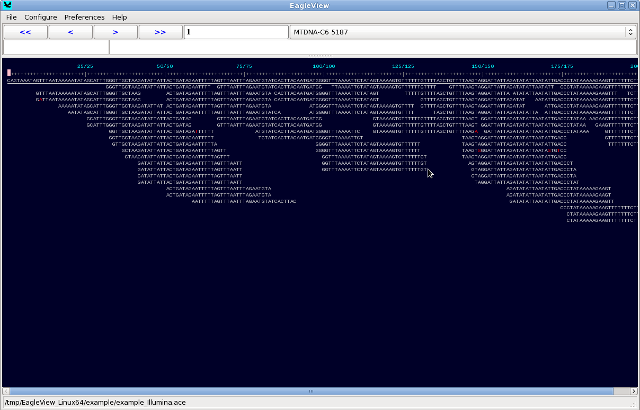 Első versenyzőnk az EagleView. A programot bináris formában terjesztik, Windows, Linux és MacOSX-re egyaránt letölthető. C++-ban fejlesztették. Akadémiai használatra ingyenes, letölteni regisztráció után lehet. A szerzők szerint még tartalmaz hibákat, amit meg tudok erősíteni. Az Overlapping Size opcióra klikkelve mindig lefagy. Funkció nem sok van benne, tudunk jobbra és balra menni a szekvenciákon. Támogatott fájlformátum egyedül az ACE. Szerintem ezt csak akkor használjuk, ha egy börtön mélyén pisztolyt nyomnak a fejünkhöz.
Első versenyzőnk az EagleView. A programot bináris formában terjesztik, Windows, Linux és MacOSX-re egyaránt letölthető. C++-ban fejlesztették. Akadémiai használatra ingyenes, letölteni regisztráció után lehet. A szerzők szerint még tartalmaz hibákat, amit meg tudok erősíteni. Az Overlapping Size opcióra klikkelve mindig lefagy. Funkció nem sok van benne, tudunk jobbra és balra menni a szekvenciákon. Támogatott fájlformátum egyedül az ACE. Szerintem ezt csak akkor használjuk, ha egy börtön mélyén pisztolyt nyomnak a fejünkhöz.
CLCBio Workbench
A CLC-ről már többször írtam, de még nem részleteztem a megjelenítőjét. A CLCBio Genomics Workbench kereskedelmi termék, 14 napos próbaidővel. Az idő lejárta után sok funkció nem érhető el, de nézegetőnek még használhatjuk. Ingyenes alternatívája a CLC Sequence Viewer, de ez nem képes megnyitni az általunk vizsgált fájlokat.
A program előnye, hogy kereshető benne az adott read neve, referencia szekvencia pozíció. Egyszerűbb szerkesztő funkciók is elérhetőek, mint amilyen az illesztés egy részletének kivágása, elmentése. Többféle formátumot ismer, a referenciára több annotációt is ráhelyezhetünk, ami hasznos, ha funkcionális összefüggéseket keresünk. Egyik nagy hátránya, hogy rendkívül ronda, a másik a readek rendezése, amit igyekszik mindig átrendezni, akárhányszor léptetjük az ablakot. Aki igényli az integrált környezetet, sok pénze van, annak jó választás lehet.
Geneious
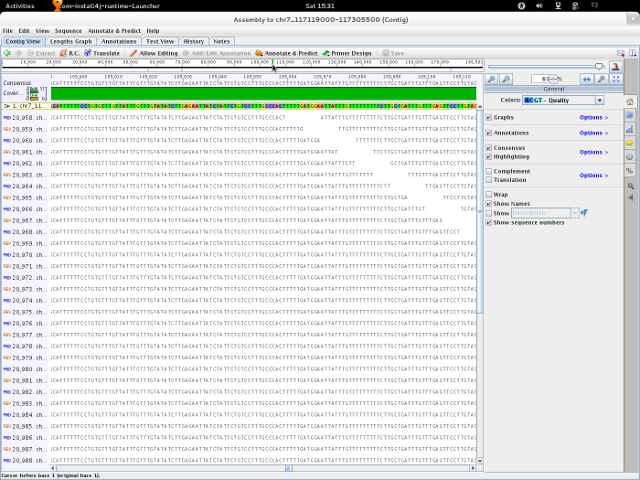 A másik kereskedelmi termék a Geneious, amiről már szintén írtam. Ott az eszközök hiányára panaszkodtam, de mi a helyzet vele, mint illesztés megjelenítővel? A felhasználói felület gyönyörű. A basic verziót ingyen használhatjuk, míg az eszközökkel megpakolt termékért fizetni kell. A Linux, Windows, MacOSX verziók mellett Solarisra is elérhető. Java nyelven írták.
A másik kereskedelmi termék a Geneious, amiről már szintén írtam. Ott az eszközök hiányára panaszkodtam, de mi a helyzet vele, mint illesztés megjelenítővel? A felhasználói felület gyönyörű. A basic verziót ingyen használhatjuk, míg az eszközökkel megpakolt termékért fizetni kell. A Linux, Windows, MacOSX verziók mellett Solarisra is elérhető. Java nyelven írták.
Az általam kipróbált példány Fedora 16-on a menüből indítva hibával kilép, de ha belépek a telepítés helyére és onnan indítom, akkor szépen fut.
A kívánt pozíciót gyorsan megkereshetjük az ablak tetején található koordináta segítségével, de akár a nevük alapján is rátalálhatunk a readekre. A menüket egérrel nem tudtam elérni, ami lehet, hogy csak a Fedora 16 egyik sajátossága, de akkor is zavaró volt. Akik nem elégszenek meg a CLC puritán kinézetével, azok nyugodtan válasszák a Geneious-t.
Tablet
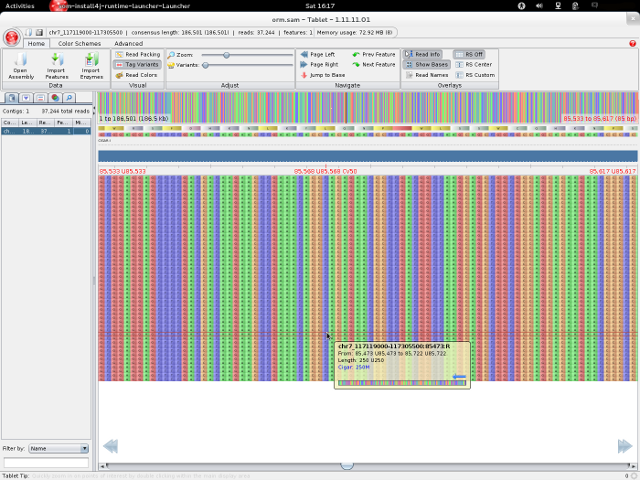 Az első program, amivel átléptünk az ingyenes alkalmazások világába, a Tablet. Java nyelven készült, elérhető az összes platformra. Ami nagyon tetszik a Tabletben, hogy több színezési módszerrel emelhetjük ki a nekünk tetsző részletet. Hátránya, hogy a navigálás primitív módon valósult meg. Nem tudunk adott koordinátára ugrani, ami például a humán 1-es kromoszómán igen nehézkessé teszi. ACE, AFG. MAQ, SAM, BAM, SOAP állományokat képes megnyitni, ez elég nagy szabadságot ad. GFF3-ban képes annotációt is betölteni. Tetszik ellenben, hogy kapunk egy áttekintő képet. Automatikusan keresi a frissítéseket, de a felhasználóra bízza, hogy az le akarja-e tölteni vagy telepíteni. Ez is szimpatikus tulajdonsága. Hátránya, hogy semmilyen szerkesztő funckiót nem kapunk. A programot csak az illesztés megtekintésére használhatjuk. Ha ezek nélkül tudunk élni, a Tablet egy remek választás.
Az első program, amivel átléptünk az ingyenes alkalmazások világába, a Tablet. Java nyelven készült, elérhető az összes platformra. Ami nagyon tetszik a Tabletben, hogy több színezési módszerrel emelhetjük ki a nekünk tetsző részletet. Hátránya, hogy a navigálás primitív módon valósult meg. Nem tudunk adott koordinátára ugrani, ami például a humán 1-es kromoszómán igen nehézkessé teszi. ACE, AFG. MAQ, SAM, BAM, SOAP állományokat képes megnyitni, ez elég nagy szabadságot ad. GFF3-ban képes annotációt is betölteni. Tetszik ellenben, hogy kapunk egy áttekintő képet. Automatikusan keresi a frissítéseket, de a felhasználóra bízza, hogy az le akarja-e tölteni vagy telepíteni. Ez is szimpatikus tulajdonsága. Hátránya, hogy semmilyen szerkesztő funckiót nem kapunk. A programot csak az illesztés megtekintésére használhatjuk. Ha ezek nélkül tudunk élni, a Tablet egy remek választás.
IGV
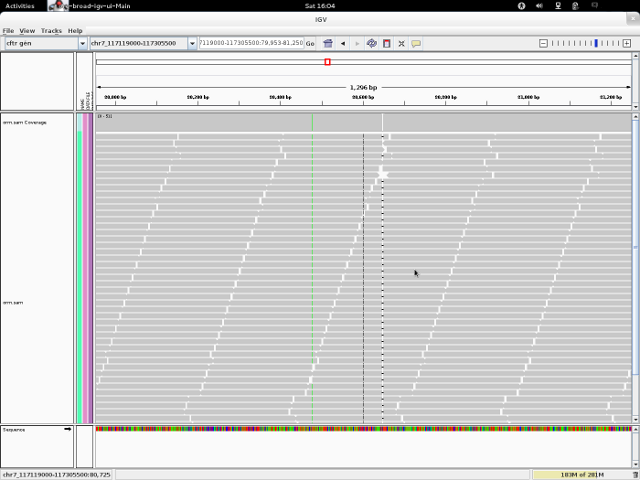 Elérkeztünk a nézegető programok leghíresebbjéhez, az IGV-hez. Egyes cégek, akik pénzért feldolgozzák a nyers szekvenciákat, képesek IGV-t csomagolni az elemzések mellé, hogy a felhasználó láthassa, mit kapott. Azt gondolhatnánk, hogy bizonyára nagyon szép felhasználói felülettel látták el. Nem. Ez a Java nyelven írt program bizony nagyon ronda. Cserébe viszont sok hasznos funkciót kapunk.
Elérkeztünk a nézegető programok leghíresebbjéhez, az IGV-hez. Egyes cégek, akik pénzért feldolgozzák a nyers szekvenciákat, képesek IGV-t csomagolni az elemzések mellé, hogy a felhasználó láthassa, mit kapott. Azt gondolhatnánk, hogy bizonyára nagyon szép felhasználói felülettel látták el. Nem. Ez a Java nyelven írt program bizony nagyon ronda. Cserébe viszont sok hasznos funkciót kapunk.
Az egyik ezek közül az igvtools, ami segítségével a sam/bam állományokon hajthatunk végre egyszerűbb feladatokat, mint az indexelés vagy sorba rendezés. Ez akkor lehet hasznos, ha a SAM fájlunk nincs koordináták szerint sorba rendezve, és ezt az IGV szóvá teszi. Anélkül, hogy a programból kilépnénk, IGV-kompatibilis állományokat hozhatunk létre. Természetesen az igazi az lenne, ha ezeket a feladatokat automatikusan oldaná meg az IGV, de ne legyünk telhetetlenek. A program hátránya, hogy nincs áttekintő kép, ami miatt először nem is látjuk, hogy hol van és hol nincs lefedettség. Ehhez nagyítani kell. Hosszú szekvenciák esetén ez hátrány, mert sokat kell lépkedni, hogy megtaláljuk azokat a régiókat, ahol nincs lefedettség.
Az IGV-t azoknak ajánlom, akik több illesztést hasonlítanak össze. Ez az eszköz az egyetlen, amelyik egyetlen referenciához több SAM/BAM állományt is be tud tölteni.
Frissítés:
Időközben találtam egy érdekes oldalt, ahol rengeteg vizualizációs eszközt lehet találni:
http://ngslib.i-med.ac.at/taxonomy/term/18
Frissítés 2:
GoldenHelix GenomeBrowser
Nemrég ingyenessé vált a GoldenHelix nézegetője is. A program C++-ban készült, Qt felhasználásával. Nem sűrűn találkozni céggel, akik keresztplatformos C++ programot fejlesztenek (Windows, Linux és MacOSX-re is kiadták, 32 és 64 bites verzióban egyaránt)! Kicsit fanyar szájízt ad, hogy használni csak regisztráció után lehet, ahol összegyűjtik az e-mailt, telefonszámot, stb.
Előnye, hogy rengeteg annotációt képes letölteni a webról. Viszont lokális gépről csak az illesztést képes betölteni, azt is BAM formátumban. Nem mertem kipróbálni, hogy nem sorba rendezett, index nélküli BAM-ot is megnyit-e, valószínűleg nem. A rendkívül szellős dokumentáció szerint IDF állományokat is meg tud nyitni, ami annotációkat tartalmazhat. Szinték képes rá, hogy több BAM-ot is megnyisson egyszerre.
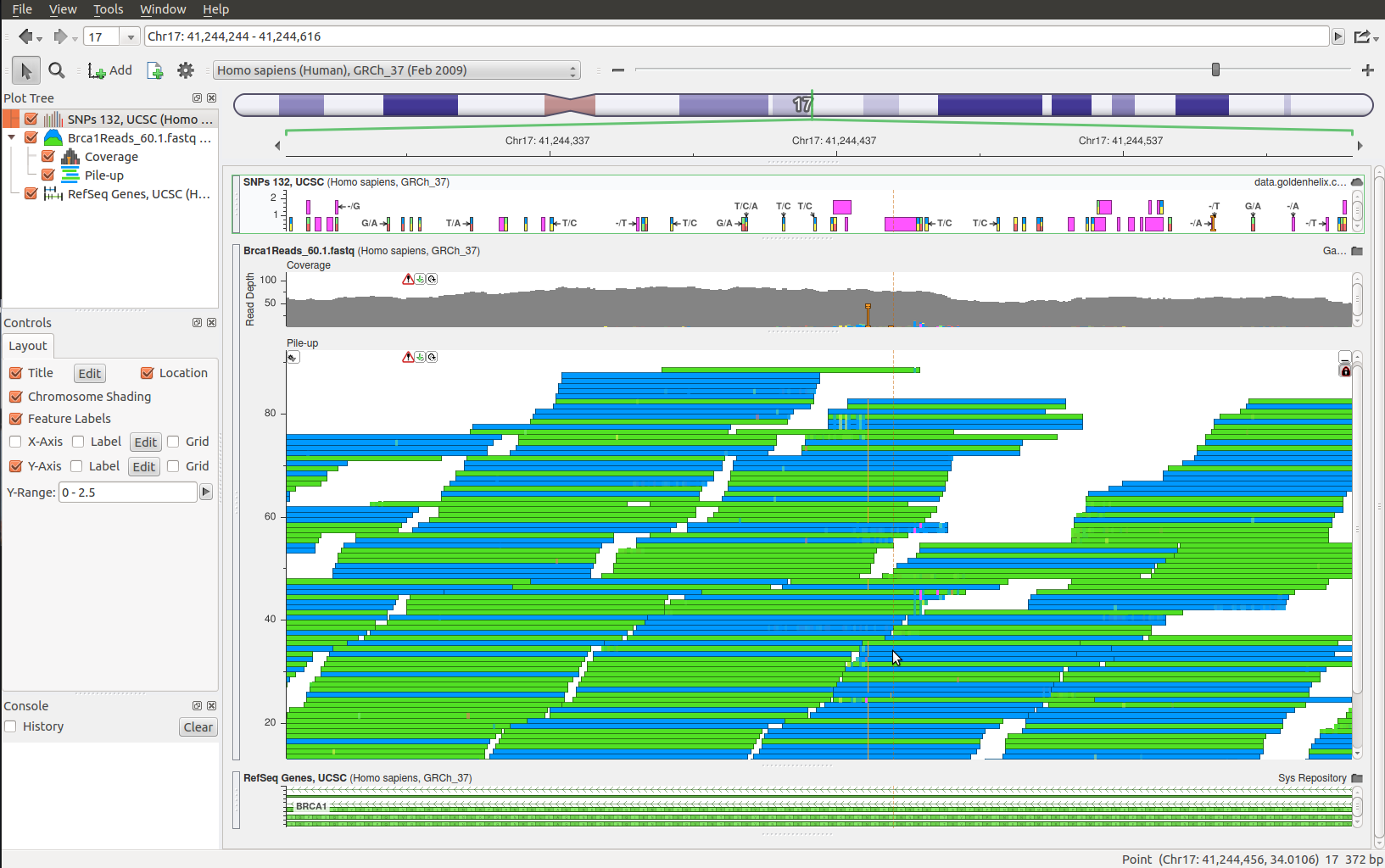
A felület nagyon tetszetős, az indexelt letöltésnek köszönhetően gyorsan megjelenít mindent, de elég kevés funkció van benne. Azoknak ajánlom, akik csak BAM fájlokkal dolgoznak és sokféle annotációt használnak, de lusták letölteni azokat.
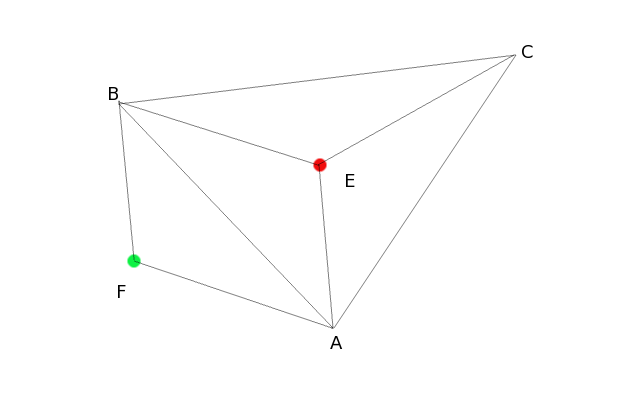 Ha megvizsgáljuk az AEB és az AFB háromszögeket, láthatjuk, hogy más a körüljárási irányuk. Az algoritmus tesztelésére készítettem egy C#-os programot, ahova idővel további triangulációs algoritmusokat is be fogok építeni.
Ha megvizsgáljuk az AEB és az AFB háromszögeket, láthatjuk, hogy más a körüljárási irányuk. Az algoritmus tesztelésére készítettem egy C#-os programot, ahova idővel további triangulációs algoritmusokat is be fogok építeni. Első versenyzőnk az
Első versenyzőnk az  A másik kereskedelmi termék a
A másik kereskedelmi termék a  Az első program, amivel átléptünk az ingyenes alkalmazások világába, a
Az első program, amivel átléptünk az ingyenes alkalmazások világába, a  Elérkeztünk a nézegető programok leghíresebbjéhez, az
Elérkeztünk a nézegető programok leghíresebbjéhez, az 